こんにちは、すきとほる疫学徒です。
R Tipsでは、私自身の備忘録も兼ねて、おすすめのRパッケージを紹介します。
80%くらいは備忘録が目的なので、殴り書きになりますことをお許しください。
tableone
複数の対象から集めたデータを使って論文を書くとき、あなたはどのようなTableを論文に入れ込みますか?
研究分野、手法によって入れ込まれるTableは千差万別だと思いますが、どのような分野、手法の研究でも「このTableだけは必ず入れる」というものがあるのではないでしょうか?
そう、収集された対象の背景情報の要約情報、いわゆるTable1というものですね。
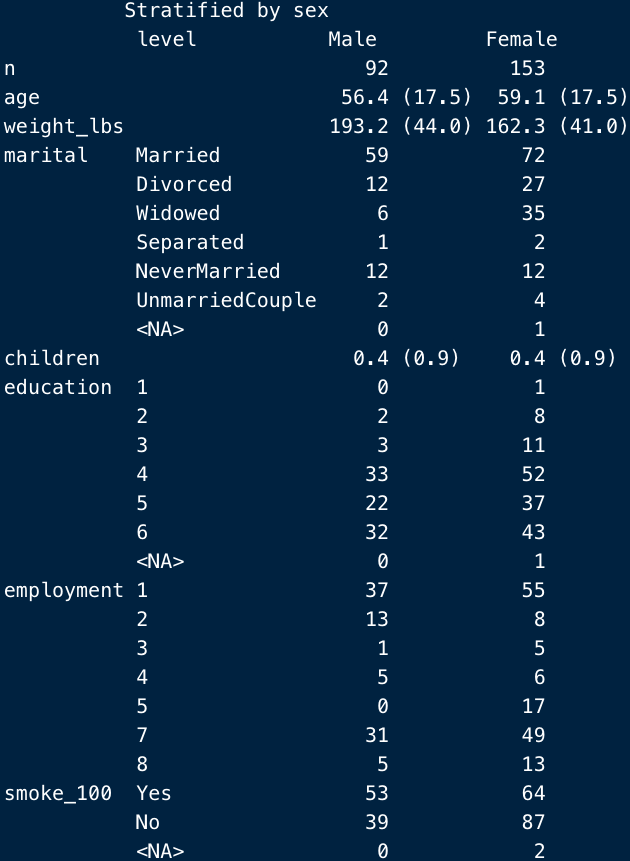
例えばこんな感じ。
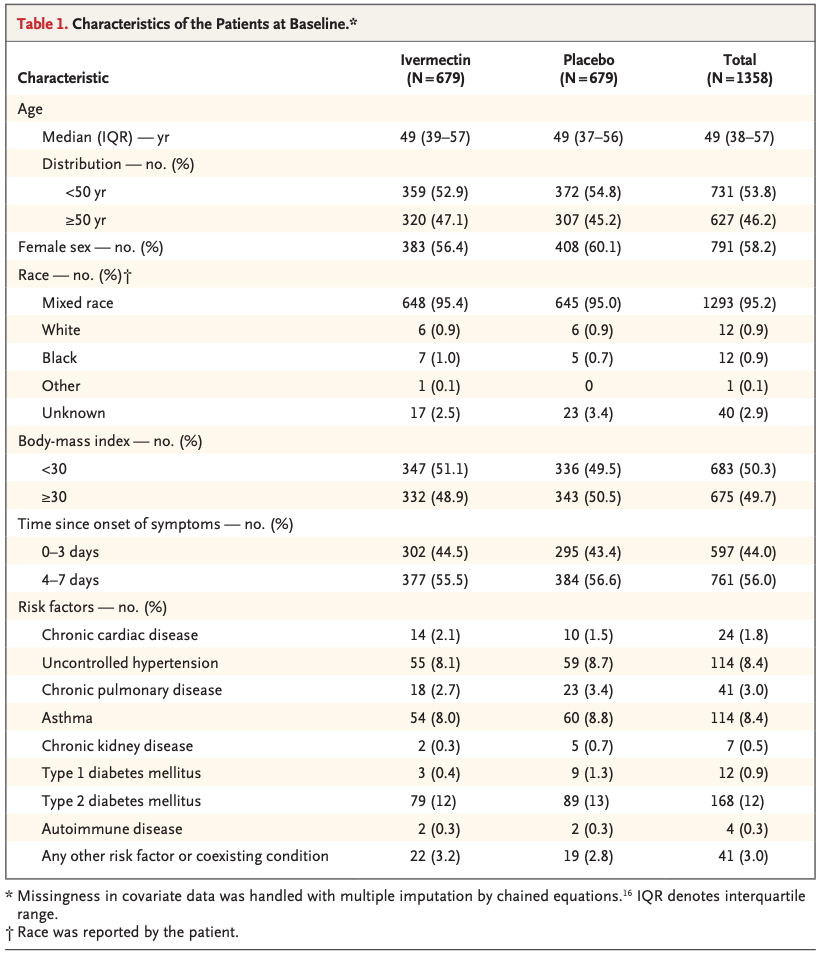
比較研究の場合は、上記のように2群+Overallの対象者背景が要約されます。
余談ですが、疫学研究におけるイケてるTable1の書き方は、以下の論文で丁寧に解説されているので、おすすめです(この論文で初めて、「あ、Table 1って、通称Table 1なんだ」と知りました)。

さて、このTable1ですが、みなさんどうやって作成していますか?
色々な方法があるかもしれませんが、それぞれの変数ごとに分布を確かめて、検定をして、その結果を手入力でExcelに入れていく、なんて方法はしんどいですよね。
時間がかかりますし、何より手入力によりヒューマンエラーのリスクが上がりますので、そのような方法は絶対に避けるべきだと思います。
そこで今回は、Table1を一瞬で作成してくれて、そのままExcelの形でデータ抽出も行ってくれる神パッケージ、tableoneをご紹介したいと思います。
なお、tableoneのオフィシャルな解説資料はこちらですので、後ほどご覧ください。
CreateTableOne()
まず、
install.packages("tableone")
library(tableone)でtableoneパッケージをインストール、読み込みましょう。
それができたら以下のようにコードを入力するだけです。
data(riskfactors)
listvars <- c("age", "weight_lbs", "marital", "children", "education", "employment", "smoke_100")
listcat <- c("marital", "education", "smoke_100")
Table1 <- CreateTableOne(vars = listvars,
factorVars = listcat,
includeNA = T,
addOverall = T,
smd = T,
data = riskfactors,
testExact = fisher.test,
testNonNormal = kruskal.test,
strata = "sex")
まず、listvars, listcatにてTable1に入れ込みたい変数群を指定します。
listvarsではすべての変数を、
listcatではそのうちカテゴリー変数だけを指定します。
ここが間違いが発生しやすい第一ポイントです。
変数を入れ忘れたり、変数名を間違えたり、連続変数なのに間違ってlistcatに追加してしまったり、カテゴリー変数なのにlistcatに入れ忘れてしまったり、ですね。
CreateTableOneでは、連続変数とカテゴリー変数に対して異なる処理を加えますので、ここはしっかりと分類してあげましょう。
それが終わったら、先ほども紹介した以下のコードで、いよいよTable1を作成していきます。
Table1 <- CreateTableOne(vars = listvars,
factorVars = listcat,
includeNA = T,
addOverall = T,
smd = T,
data = riskfactors,
testExact = fisher.test,
testNonNormal = kruskal.test,
strata = "sex")
引き数を順に説明します。
vars/factorVars
先ほど作成した全変数、カテゴリー変数群のリストを、ここで指定します。
varsには全変数のリストを、
factorVarsにはカテゴリー変数のリストを指定しましょう。
ここもよく間違いがあるポイントで、作成されたTable1がなんかおかしいと思ったら、全変数とカテゴリー変数のリストを逆に指定してしまっていたということが稀にあります(私がおっちょこちょいなだけですね💦)
includeNA
includeNAは、各変数ごとに欠損のカテゴリーを表示するかどうかのオプションです。
デフォルトでは表示なしになっているため、もし表示したければ、
includeNA = Trueを指定してあげる必要があります。
Table1には欠損の頻度、分布を把握するという役割がありますので、ここはTrueにしてあげた方が良いでしょう。
こちらがincludeNA = False (デフォルトですので、入力の必要なし)
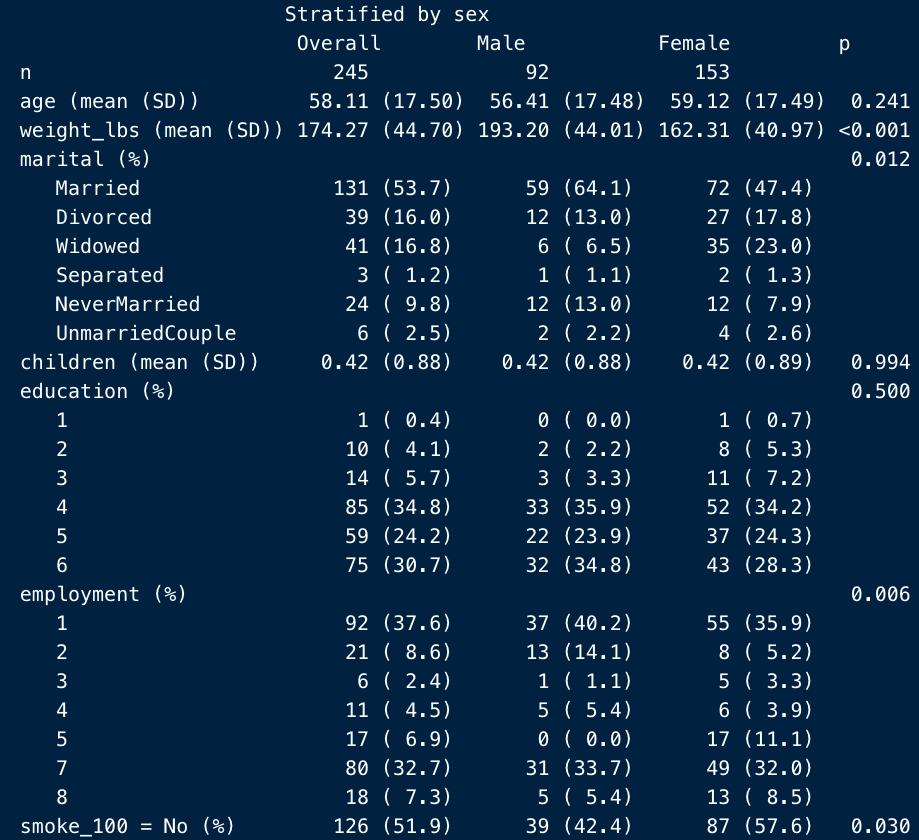
こっちがincludeNA = Trueです。
ご覧のように、各変数にNAというカテゴリーが追加されています。
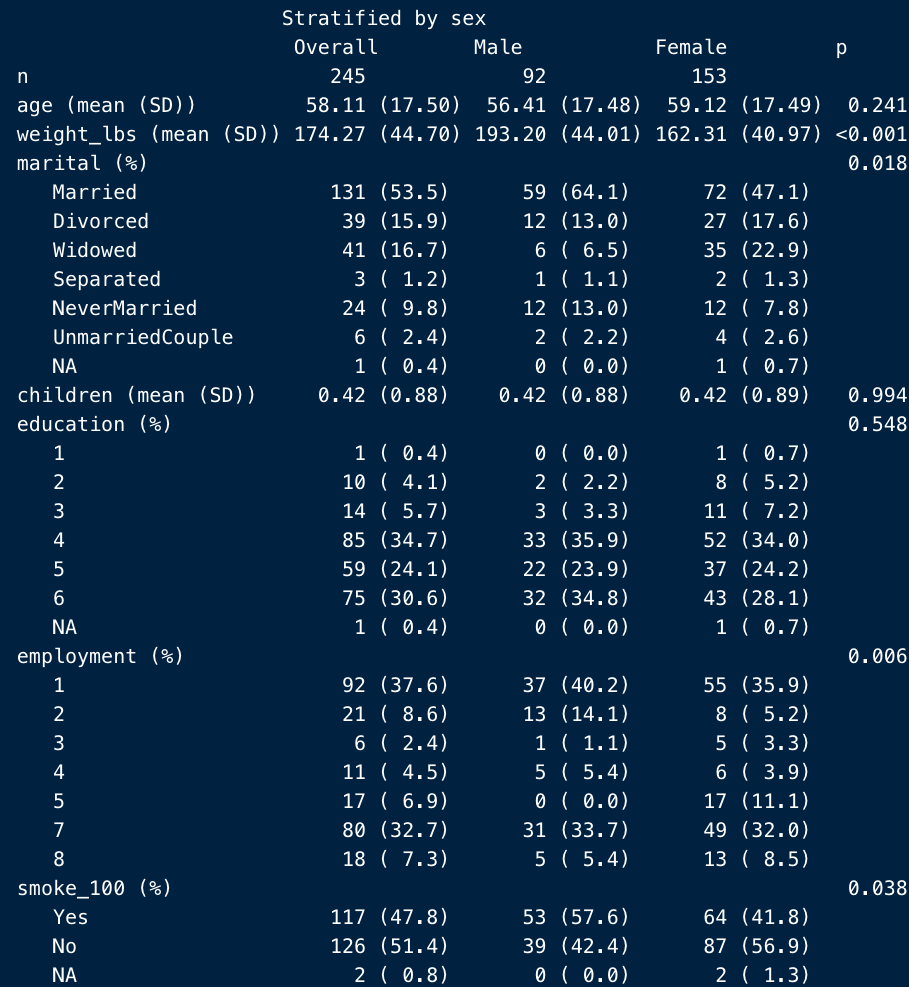
addOverall
addOverallは、何らかの変数(今回は性別)で層別化されたTableを作成する際に、それに加えてOverallの列を追加するかどうかを指定する引き数です。
デフォルトでは追加しないようになっています。
addOverall = False(デフォルトなので、入力の必要なし)
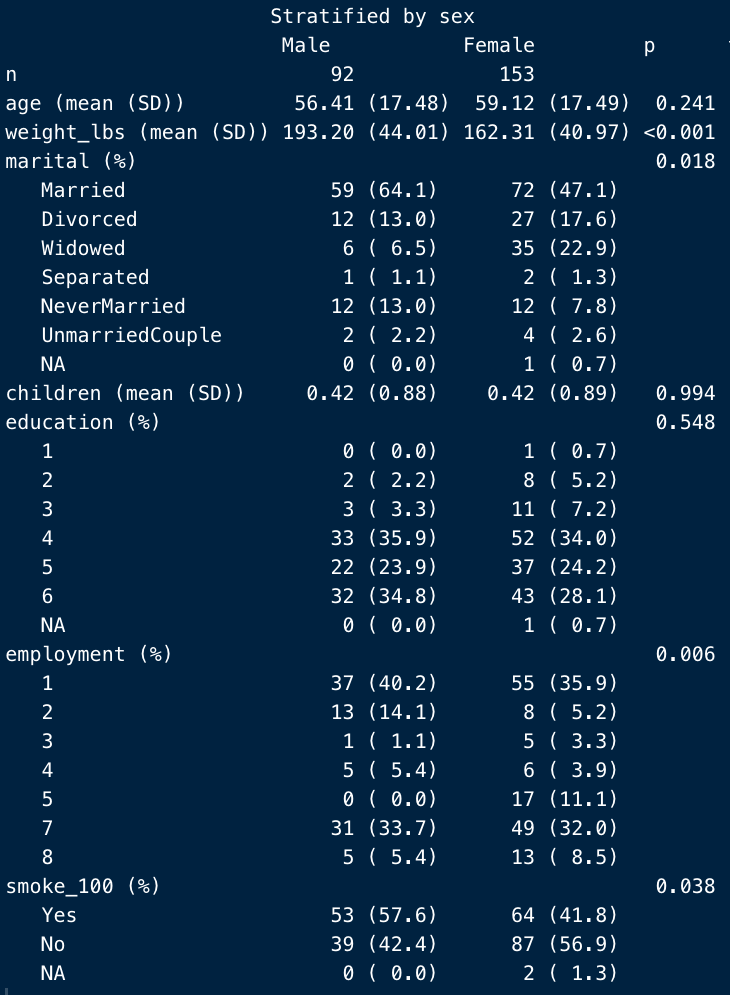
addOverall = True
以下の通り、左にOverallという列が追加されていますね。

strata/data
strataは層別化したい変数を、データはTable1を作成する元となるデータを指定します。
strataを指定しなければ、単にOverallでの変数分布が表示されます。
smd, testExact, testNonNormal
smdはStandardized mean differenceを表示するかどうかのオプションです。
傾向スコアによるバランシングを行った際に、両群のバランスをチェックしたい時などには入れることが多いですね。
こちらのCreateTableOne()は、大変便利なことに自動で各変数の2群間での分布に対して検定を行ってくれます。
何も指定しない場合は、連続変数に対してはT検定が、カテゴリー変数に対してはカイ二乗検定が行われます。
もし上記以外の検定を行いたい場合には、
testNonNormal = kruskal.test
とすることで、連続変数に対してはマンホイットニーのU検定が、
testExact = fisher.test
とすることで、カテゴリー変数に対してはフィッシャーの正確検定が実施できます。
print()
上記工程により、Table1を作成することができます。
これをExcelで吐き出すためには、print()に通してやる必要があるのですが、この段階でも引き数によりTable1をデコレートすることができるので、ご紹介します。
全体のコードはこんな感じ。
print(Table1,
showAllLevels = T,
smd = T,
explain = F,
catDigits = 1,
contDigits = 1,
pDigits = 2,
test = F,
format = "pf") %>%
write.csv("/Users/***/Desktop/R_file/Test/Table1.csv")
showAllLevels
CreateTableOneでは、2値のカテゴリー変数に対しては、
Sex=Female のように、どちらか1群の要約情報しか表示されません。
それに対して、Female/Maleの双方のようやく情報をTableに入れ込みたい場合には、
showAllLevels = True
を指定してやりましょう。
こちらの方が、Table1を見た読者に対して「この変数にはどんなカテゴリーがあるか」ということがクリアに伝わるため、基本はshowAllLevelsはTrueにした方が良いと思います。
explain
CreateTableOneは、以下のように各変数の名前の後に、%やmeanなど、どのような指標で要約しているかが記載されています。
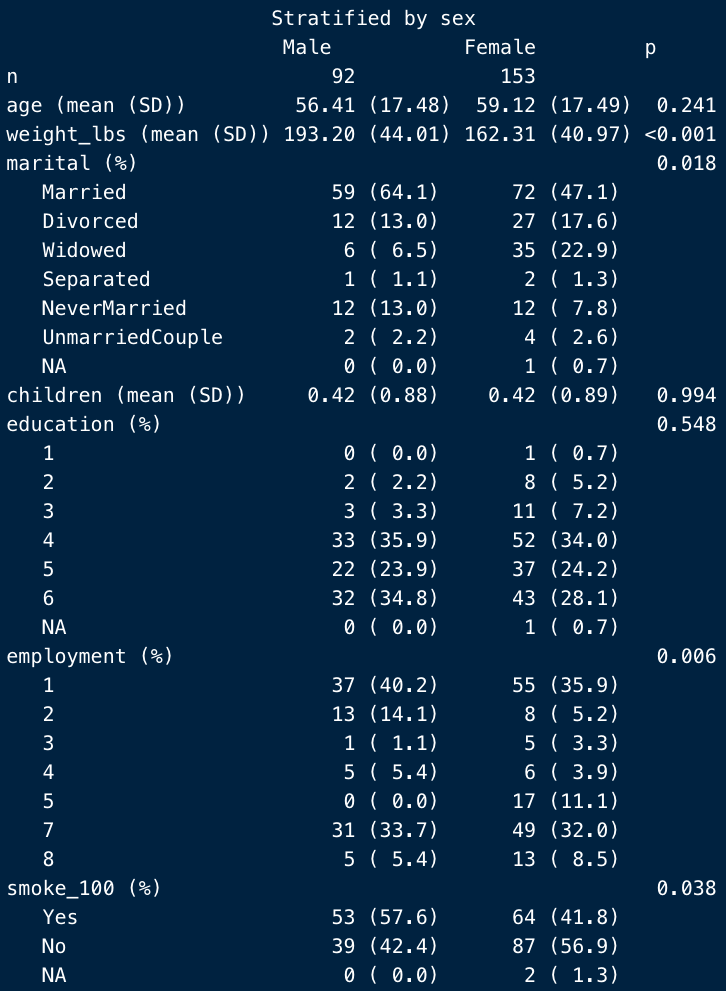
もしこれが鬱陶しいと感じるのであれば、
explain = False
を指定してやれば、以下のように消すことができます。
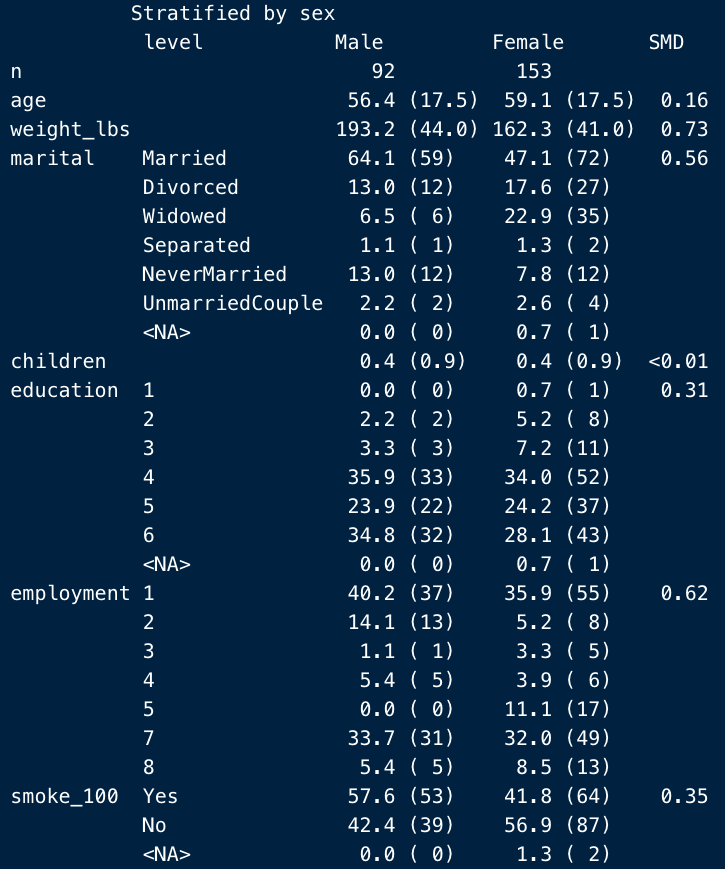
ただ、個人的には%やmeanという指標を見ることで、読者も「あ、連続値ね、カテゴリーね」とスムーズに変数のタイプを識別することができますので、基本はここはいじらない方が良いと思います。
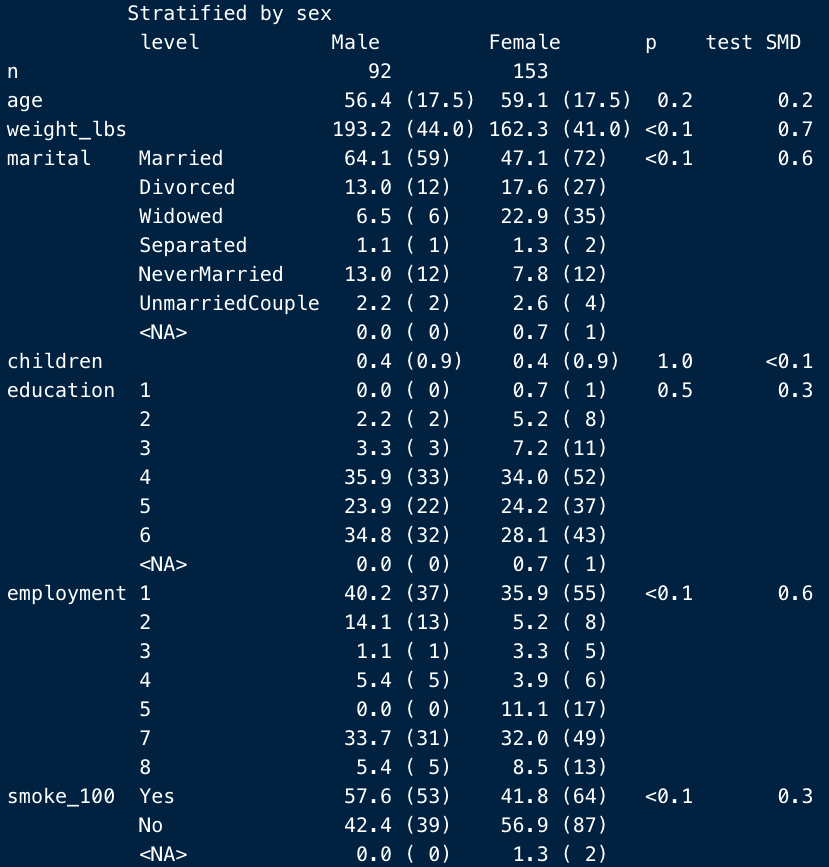
catDigits, contDigits, pDigits
これは、要約指標として表示される頻度、mean、%およびP値/SMDを小数点何桁まで表示するかという指示です。
一桁を指定すれば、こう
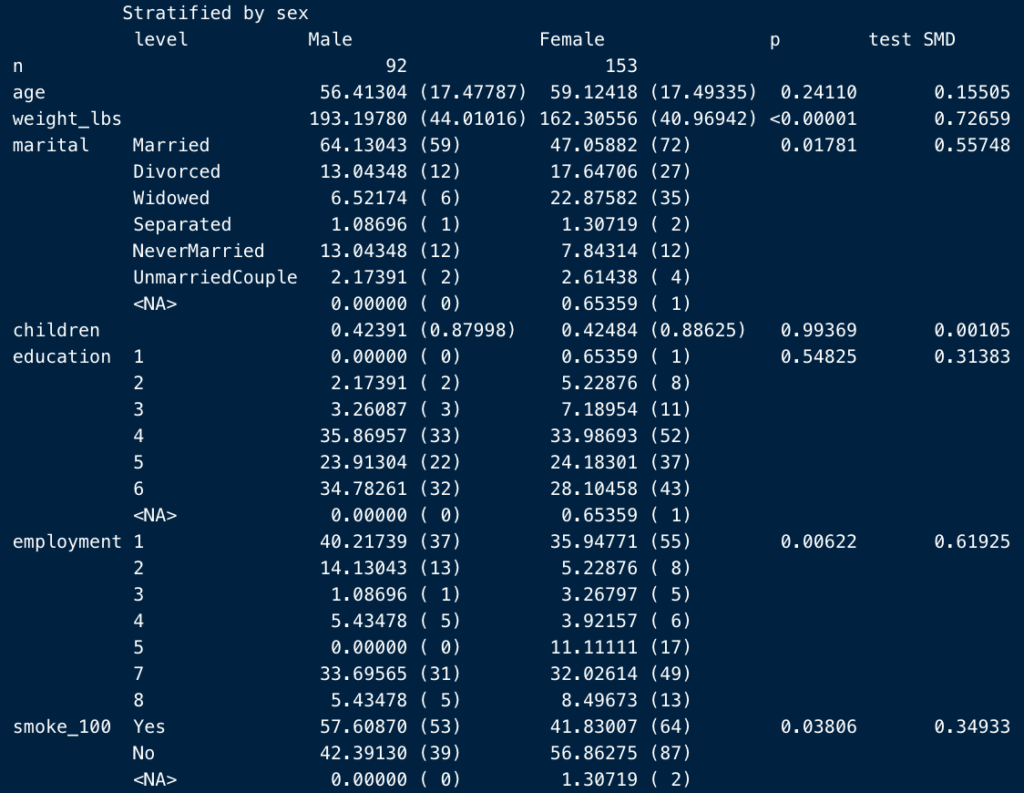
極端に5桁を指定すると、こうですね。
test
Table1においては、「P値を掲載する必要はない(してはならない)」という主張をするJournalもありますし、私も個人的には掲載するべきではないと考えています。
そんなときは、
test = False
を指定してやれば、P値そのものをTableから消すことができます。
こんな感じ。
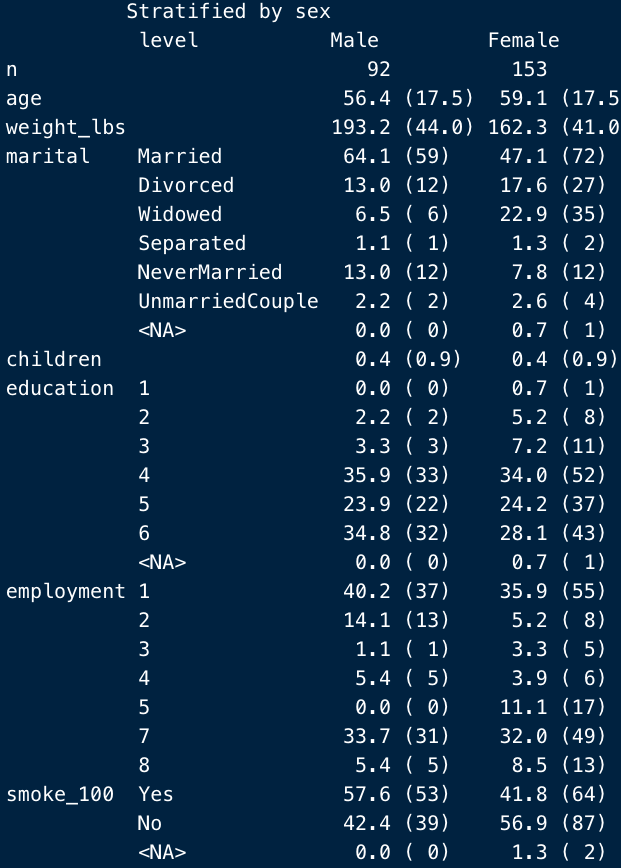
format
最後に、formatでは、表示する要約指標とその順番を選択することができます。
デフォルトでは、以下のように頻度(%)が表示されます。
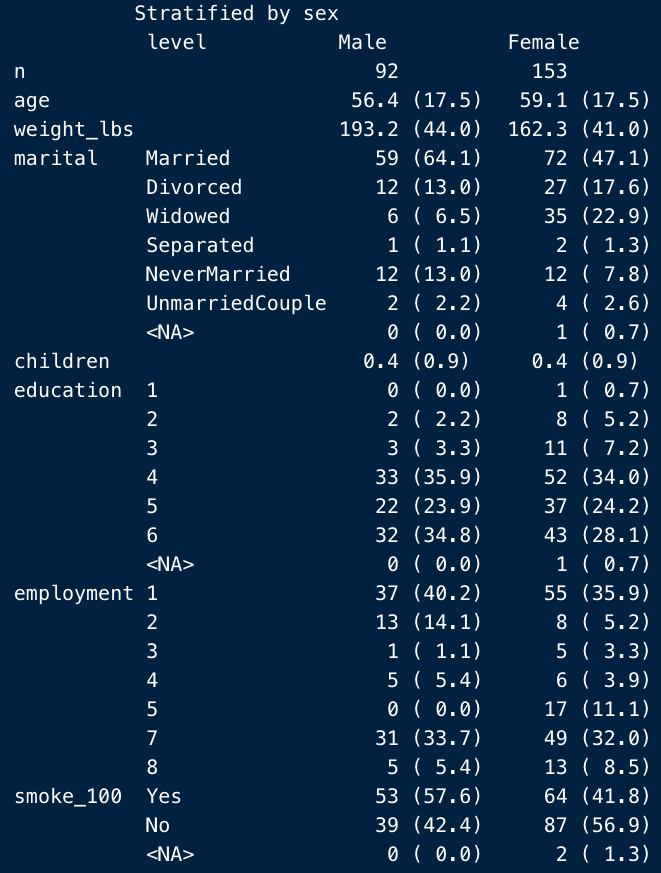
これに対し、format = “pf”なら、%(頻度)
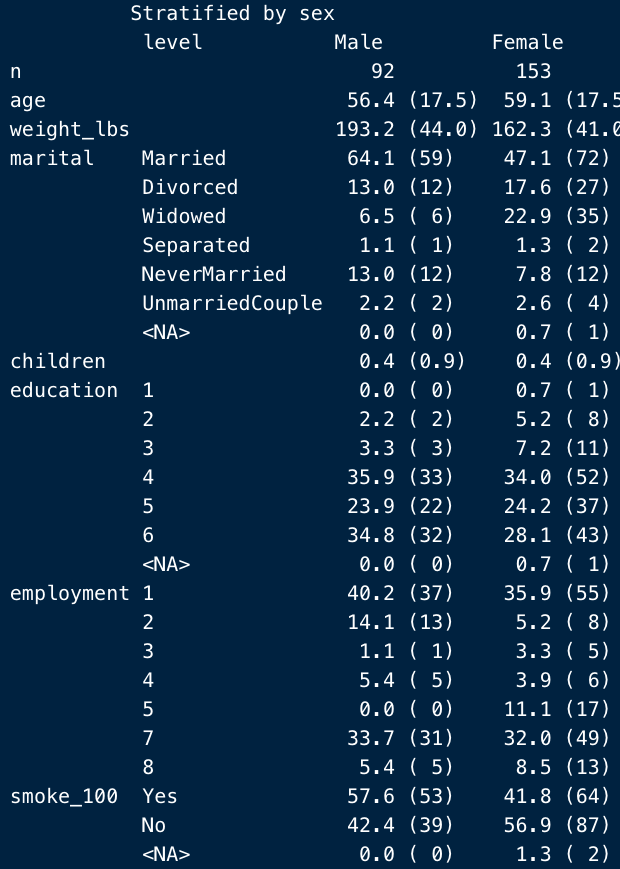
これに対し、format = “p”なら、%のみ
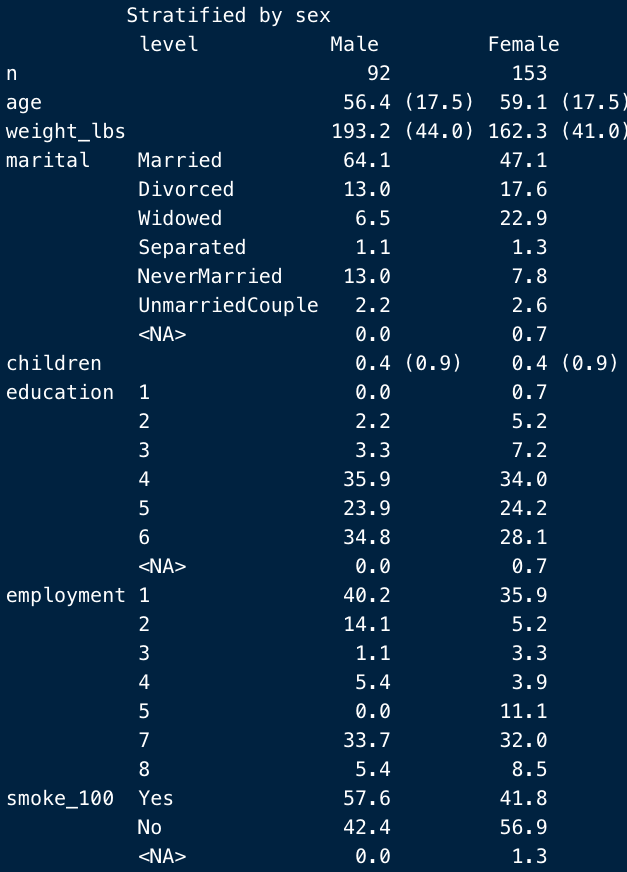
これに対し、format = “f”なら、頻度のみです。
write.csv()
上記のようにprint()でTableの表示形式を選択するだけでは、excelに吐き出すことはできません。
このように、write.csv()に渡してあげることで、初めて指定したパスにexcelファイルが保存されます。
print(Table1,
showAllLevels = T,
smd = T,
explain = F,
catDigits = 1,
contDigits = 1,
pDigits = 2,
test = F,
format = "pf") %>%
write.csv("/Users/***/Desktop/R_file/Test/Table1.csv")
ちなみに、(私もめっちゃやるのですが)、パスの最後で指定するファイル名に、”.csv”をつけないと、excel形式にならないので注意しましょう。
以上で、tableoneパッケージの説明は終わりです。
すきとほる疫学徒からのお願い
本ブログは、読者の方が自由に記事の金額を決められるPay What You Want方式を採用しています。
「勉強になった!」、「次も読みたい!」と本ブログに価値を感じてくださった場合は、以下のボタンをクリックし、ご自身が感じた価値に見合うだけの寄付を頂戴できますと幸いです。
もちろん価値を感じなかった方、また学生さんなど金銭的に厳しい状況にある方からのご寄付は不要です。
引き続き情報発信していく活力になりますので、ぜひお気持ちに反しない範囲でご寄付をお願い致します!








