こんにちは、すきとほる疫学徒です。
【初心者のためのRで医療ビックデータ解析】シリーズは、「これまでデータの解析を行ったことがない人」を対象にして、Rで医療ビックデータを解析する方法をわかりやすく解説していきます。
全てのシリーズを受講し終えた時点で、「診療報酬データやレジストリーといった医療大規模データを、自立して解析できる」状態になることを目指しております。
全研究者、Rユーザー化計画、ここに始動す。
こちらの記事はパート5になりますので、まずそれ以前のパートをお読み頂いてから、この先に進んで頂けますと、より一層Rの勉強が楽しめるかと思われます。
変数作成におけるTips
この章では、Rを使って解析に必要な変数を作成する方法を学んでいきます。
その上で押さえておくべきTipsがあるので、最初にさらっていきましょう。
変数名は全て英語、かつ記号は_のみ
以前の記事でお伝えした通り、Rでは日本語がうまく作動しません。
ですので、新たに変数を作成する際には必ず英語を使いましょう。
また、同じ理由でほとんどの記号が作動せず、使えるのは”_”のみです。
変数名は上書きしない
例えば、連続変数であるageを加工して、カテゴリー変数のageを作るとしましょう。
この際、作成する変数名は元のageという変数名とは異なる変数名にしなければなりません。
理由は二つです。
一つ目は以下で紹介した通り、コードエラーのダブルチェックをするためです。
Codeを書いてageという変数を加工しても、本当にあなたが望む通りに加工できているかはしっかりと確認しなければわかりません。
ですので、変数作成後には必ず、元の変数と新しい変数を比較し、想定した通りの加工が行われているかをチェックしましょう。
このために、元の変数を残しておく必要があります。
二つ目は、修正をしやすくしておくためです。
例えば、
「ageのカテゴリーを2つにしたけれど、やはり3つにしたい」
「そもそもカテゴリー変数ではなく、連続変数として解析に投入したい」
と修正が必要な状況になったとしましょう。
この時、元の変数を残しておけば、少しコードをいじるだけですぐに修正を反映できます。
一方で、元の変数が完全に消えてしまっている場合は、ひどい時にはデータ全体を再度読み込みなおなねばならないことがあり、データの容量が大きい場合には手間になります。
これら二つの理由から、変数を加工する時にも、必ず元の変数は残しておくようにしましょう。
ただ、そのような変数がいつまでもデータセットに残り続けているのも解析に誤った変数を投入してしまうリスクをあげますので、私はいつも解析の直前に不要な変数を全て落とし、解析に必要な変数だけを含んだデータフレームを作成するようにしています。
変数名に規則性をもたせる
というわけで、Rで変数を加工していくと、続々と新しい変数がデータフレームに追加されていくことになります。
ですので、「この変数は、こういった加工を施した変数!」ということを、変数名を見ただけで一瞬で識別できるような、自分だけの規則を作るようにしましょう。
何度もRを使って解析していると、「あー、この名前だと混乱しやすいな、ああしてみよう」というように、自分でお気に入りの名付け方が見えてくると思うので、試行錯誤してみてください。
これは変数名ではなく、作成したデータフレームや、解析結果にも言えることです(Rでは、ロジスティック回帰などの解析結果にも名前を与えて、保存します)。
例えば私の場合は、変数名なら
- カテゴリー変数は最後に”_c”をつける
- 冒頭も含めて全て小文字
- 最新の変数がわかるように、変数名の後にナンバリングする
というような感じです。
ageに対し、3度ほど加工し、最終的にカテゴリー変数になった場合には、
age3_cといった名前になるということですね。
データフレームの場合は、
- 最初に読み込んだ際には”df”とする
- 対象の包含・除外基準を適用するにつれて、変数名の後にナンバリングする
- 解析対象のデータフレームが作成できたら、”dfx”とする
- 感度分析・追加解析を行う際には、”dfx_”で、_の後に感度分析・追加解析の内容がわかるワードを追加する
というような感じです。
例えば、3つの包含基準、2つの除外基準を適用させて、
df5となり、
そこで解析対象がFixであれば、
dfxとします。
さらに、たとえば解析対象を年齢別にサブグループ解析する時には、
dfx_agelow
dfx_agemid
dfx_agehighとします。
追加解析・感度分析の数が10を超えてくると、「誤ったデータフレーム名を書いてしまった」なんてことも全然起こりうるので、データフレームを名づける時には、一眼見て識別できる名前をつけるように心がけましょう。
また、解析結果を名づける時には、
- 冒頭は解析手法
- 次に対象集団
- そのまた次に主たる曝露(バリエーションがあれば)
- 最後にアウトカム(バリエーションがあれば)
としています。
たとえば、
一般化線形モデルを使い、
対象集団は低年齢のサブグループ、
主たる曝露が薬剤Aで、
アウトカムが在院日数、
だとすると、
"glm_agelow_drugA_LOS"というふうに名づけています。
データフレーム同様に、こちらも感度分析・追加解析が増えてくると、どの解析結果がどの解析に対応しているのか、本当に混乱しますので、一眼見て識別できる名称をつけてあげましょう。
カテゴリー変数はカテゴライズの方法がわかるようにする
たとえば、
deathという変数があり、カテゴリーが1/0だったとしましょう。
さて、1/0のどちらが死亡ありで、どちらが死亡なしですか?
「deathっていう名前の変数だから、1が死亡なんじゃない?」
良いでしょう、ではこちらは?
sexカテゴリーは同様に1/0です。
さぁ、どっちが女性でどっちが男性でしょうか?
「いや、でも自分でコーディングしてるんだから、作った時にどっちを1でどっちを0にしたかなんて覚えてるよ」と思うかもしれません。
安心してください、絶対に忘れます。
医療大規模データを解析するCodeを書く上では、自分を信用してはいけません。
仮に「死亡=1、非死亡=0」だと思っていたものが、本当は真逆だとしたら?
当然、解析の結果も真逆になりますよね?
しかし、当の解析者本人はそのことに気づかず、「薬剤Aの内服が、死亡を抑制する効果があることを示唆している‼️」などと声高に叫んでしまうかもしれません(本当は抑制どころか、死亡を誘発しているのに)。
恐ろしいですね。。。
以前も説明した通り、医療大規模データ解析においては、本当に些細なコーディングエラーが致命的なミスを引き起こします。
というわけで、カテゴリー変数を作る際には、必ず変数名からカテゴライズの法則がわかるようにしておきましょう。
deathという変数であれば、1/0ではなくyes/noとコーディングしておく、
sexという変数であれば、female/maleにする、もしくは変数名自体をfemaleにしてyes/noとしておく、
などの工夫が可能です。
変数を加工してみよう
では、上に述べた法則に則って、変数を加工していきましょう。
ここからは、変数の加工に必要な関数を紹介してきます。
mutate()
mutate()は、新たに変数を追加する際に使用する関数です。
たとえば、連続変数であるageとアウトカムのdeathとの間に非線形の関係が観察されたため、解析にageの二乗項を入れるとしましょう。
df <- df %>%
mutate(age_2 = age*age)
df %>%
select(age, age_2) %>%
head()
mutate()では、まず新たに作りたい変数名を左側に書き、=で繋いだ右側にその条件を書きます。
df <- dfとしているのは、単にdf %>% mutate()とするだけでは、瞬間的にage_2という変数を作成するだけで、age_2という変数を持ったデータフレーム自体が作成しないからです。
なので、
dfという新たな変数を作ってくださいね:df <-
それは、dfに対してmutate()を使って〜:df %>% mutate()
という指示を出しているわけですね。
ちなみにここでは、変数を追加しているだけで、包含・除外条件の適用のように対象集団自体が変わるような加工を行っていないために、df1のように新たな名称のデータフレームは作成していません。
今回はそういった変数は用意していませんが、たとえば
df <- df %>%
mutate(LOS = entday - admday + 1)
で在院日数を計算したり(entdayは退院日、admdayは入院日)、
df <- df %>%
mutate(bmi = weight/height^2)でBMIを計算したりします。
さて、そんなmutate()ですが、これから紹介するいくつかの関数と組み合わせることで、さらに複雑な変数を作成することができます。
case_when()
case_when()は、とてもとてもとてもとてもとてもよく使用します。
たとえば、ageをカテゴリー変数にしたいとすると、こんな感じ。
df <- df %>%
mutate(age_c = case_when(age <= 19 ~ "-19",
age > 19 & age <= 64 ~ "20-64",
age > 64 ~ "65-"))
df %>%
select(age, age_c) %>%
head()
mutate()で作成したい変数名を書き、=の右にその条件を書いています。
case_when()は、左側に条件を書き、~の右側にその条件を満たすカテゴリー名を書きます。
事前に「Rでは記号は”_”しか使えない「と述べていましたが、文字列のカテゴリー名を作成する際にはその他の記号やスペースも使用することができます。
ほかにも、連続変数である喫煙をカテゴリー変数にしたければ、
df <- df %>%
mutate(smoking_c = case_when(smoking_status == 0 ~ "No smoker",
smoking_status >= 1 & smoking_status <= 50 ~ "Moderate smoker",
smoking_status > 50 ~ "Severe smoker"))
df %>%
select(smoking_status, smoking_c) %>%
head()
ちなみに、Rで等式を使う際には、上のように”=”ではなく”==”と書く必要があります。
さらに、上記では&のみ使用していますが、もしA or Bのように条件づけしたければ、”|”で繋いでやればOKです。
また、case_when()の中身は連続変数だけでなく、カテゴリー変数をとることもできます。
その場合は、等式・不等式の右側にカテゴリー名を記載することになります。
では、ここで課題です。
連続変数であるADLを、以下のようなカテゴリー変数にしてみましょう。
0-10: Low ADL
11-90: Mid ADL
91-100: High ADL
いかがでしょうか?
はい、コードにするとこうですね。
df <- df %>%
mutate(adl_c = case_when(ADL <= 10 ~ "Low ADL",
ADL >= 11 & ADL <= 90 ~ "Mid ADL",
ADL <= 100 ~ "High ADL"))
df %>%
select(ADL, adl_c) %>%
head()
繰り返しになりますが、変数作成後には必ずオリジナルの変数と見比べて、求めていたカテゴリーが作成できているかを確認しましょう。
では、sexという変数をfemaleにして、女性ならYes、男性ならnoとするにはどうしたらいいでしょうか?
こうですね。
df <- df %>%
mutate(female = case_when(sex == "1" ~ "Yes",
sex == "0" ~ "No"))
if_else()
さて、case_whenと同じような関数にif_else()というものがあります。
case_whenは複数通りの条件を設定できたのに対し、if_else()は2通りまでの条件設定を行います。
たとえば、先ほどcase_when()で作成したfemaleという変数をif_else()で作成すると、こうです。
df <- df %>%
mutate(female = if_else(sex == "1", "Yes", "No", missing = NULL))
df %>% select(sex, female) %>%
head()
if_else()の中で、まず変数とその変数に対して設定する条件を書き(sex == “1”)、その次にその条件を満たした際に返す値を書き(”Yes”)、最後にそ例外の時に返す値を書きます(”No”)。
このように、「AならばX、それいがいはY」という形で条件付けを行いますので、if_else()と呼ばれているわけですね。
もちろん、それぞれの条件に対して文字列でなく数値を書くこともできます。
欠損の取り扱い
さて、上記で紹介したcase_when()とif_else()ですが、変数によっては欠損を何らかの値に置き換えたい、もしくは何らかの値を欠損に置き換えたいという状況があるかもしれません。
たとえば、オリジナルのデータで欠損が9999という数値で記載されている時などですね。
このような時も、mutate()、if_else()で対応することができます。
たとえば、連続値のADLに対して、0を欠損にしてやりたいとしましょう。
0を考慮しない場合のコードは以下でしたね。
df <- df %>%
mutate(adl_c = case_when(ADL <= 10 ~ "Low ADL",
ADL >= 11 & ADL <= 90 ~ "Mid ADL",
ADL <= 100 ~ "High ADL"))
df %>%
select(ADL, adl_c) %>%
head()
これに対し、0を欠損するという条件を加えると、
df <- df %>%
mutate(adl_c_na = case_when(ADL == 0 ~ NA_character_,
ADL >= 1 & ADL <= 10 ~ "Low ADL",
ADL >= 11 & ADL <= 90 ~ "Mid ADL",
ADL <= 100 ~ "High ADL"))
このように、case_when()の冒頭に
ADL == 0 ~ NA_character_,という行が追加されています。
余談ですが、Rには変数の方があるとお伝えした通り、欠損を表すNAにも方があります。
NA_integer_
NA_real_
Na_character_
NA_complex_
です。
上から順にそれぞれ、integer、double、character、complexに対応しています。
今回は、他のカテゴリーを文字列で認識させているので(”Low ADL”など)、作成される変数がcharacter型となります。
そのため、欠損もcharacter型である必要があるので、NA_character_を指定したわけですね。
試しに、NA_integer_を指定すると、
df <- df %>%
mutate(adl_c_na = case_when(ADL == 0 ~ NA_integer_,
ADL >= 1 & ADL <= 10 ~ "Low ADL",
ADL >= 11 & ADL <= 90 ~ "Mid ADL",
ADL <= 100 ~ "High ADL"))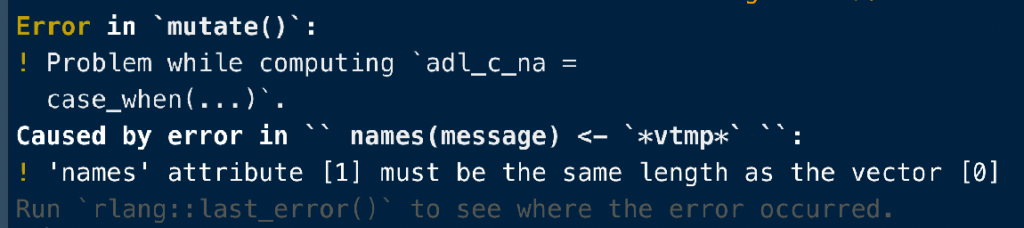
こんな感じでエラーが出ます。
もし他のカテゴリーを数値にした場合には、NA_real_を入れてやれば、解析が回ります。
df <- df %>%
mutate(adl_c_na = case_when(ADL == 0 ~ NA_real_,
ADL >= 1 & ADL <= 10 ~ 1,
ADL >= 11 & ADL <= 90 ~ 2,
ADL <= 100 ~ 3))
NAの型はうっかりしていると結構な頻度で間違えてエラーが返ってきますので、気をつけましょう。
私は作成後の変数の方を確認するのが面倒なので、エラーが返ってきた時にはとりあえずその他のNA_****の方を入れて、回ったものを使うようにしています。
across()
これはRに慣れてくれば使ってみると良いと思いますが、across()という非常に便利な関数があります。
たとえば、複数の変数に対して0/1を”No”/”Yes”に変換するような作業をするとしましょう。
そんな時に、変数ごとにこのようなコードを書くのは非常に面倒ですよね。
df <- df %>%
mutate(female = case_when(sex == "1" ~ "Yes",
sex == "0" ~ "No"))
across()は、対象に選択した複数の変数に対して、同時に指示を飛ばすことができます。
こんな感じ。
df <- df %>%
mutate(across(c(academic_hospital,
death,
dementia,
drug_A,
drug_B,
liver_disease,
myocardial_infarction,
solid_tumor),
~ case_when(. == "1" ~ "Yes",
. == "0" ~ "No")))
他にも、以下のように一括でfactor型の変数を指定することもできます。
df <- df %>%
mutate(across(where(is.factor),
~ case_when(. == "1" ~ "Yes",
. == "0" ~ "No")))ただ、このような一括選択だと、思いもよらぬ変数まで指定されてしまう可能性があるので、私は面倒でも上記のように一つ一つ変数名を書くようにしています。
変数の指定方法は他にも様々ですので、ぜひネットで検索してみてください。
ちなみに、上のようなCodeだけだと、オリジナルの変数がそのまま加工された変数に置き換わってしまい、作成前後での比較ができません。
そんな時は、以下のように”.names=”を使ってやりましょう。
df <- df %>%
mutate(across(where(is.factor),
~ case_when(. == "1" ~ "Yes",
. == "0" ~ "No"),
.names = "{.col}2"))
“”の中に新たに名付けたい変数名を書き、今回は2を付け加えています。
すると、このようにacross()で選択した変数名の後ろに2が追加されていることがわかります。

もちろんながら、2でなくとも{.col}_100とか、Anyname_{.col}_9999とか、好きなような名前をつけてあげることができます。
ordered() & relevel()
多変量回帰などの回帰分析にカテゴリー変数を投入する際には、各変数ごとにrefereneとなるカテゴリーを定めます。
また、データの記述統計量を算出する際にも、カテゴリーの並び方に従って結果が返ってきます。
たとえば、age_cという変数に対して、おそらく多くの方はLow, Mid, Highという並び順でカテゴリーを配置し、さらにreferenceにはLowを設定したいのではないでしょうか?
そんな時、もしmutate()でageをカテゴリー変数化した際に、Midがreferenceになっていたらどうするか。
そこで登場するのが、ordered()とrelevel()関数です。
ordered()は、以下のようにその変数のカテゴリーの並び順を全て指定します。
df$smoking_c <- ordered(df$smoking_c, c("No smoker", "Moderate smoker", "Severe smoker"))
すると、df$smoking_cと打ち込むと、以下のようにカテゴリーの並び順を示すLevelsという行が追加されるようになります。

Rは賢いので、連続変数をカテゴリー変数にした際には、小さい数字に対応するカテゴリーから準備levelを決めてくれるのですが、私は念の為、カテゴリー変数を作成した際には必ずlevelを再定義してやるようにしています。
relevel()も似たような関数ですが、こちらはreferenceとなるカテゴリーのみを指定します。
こちらは、
df$変数 <- relevel(df$変数, "referenceにしたいカテゴリー名")で設定できます。
group_by()
解析をしていると、グループ別(たとえば居住エリアとか)に要約した何らかの変数を作成したいというモチベーションが生まれることがあります。
医療大規模データベース研究ですと、病院ごとのオペ回数を合計したHospital volumeなどですね。
各種オペにおいて、Hospital volumeは患者の予後を予測する重要な因子であることが明らかになっておりますので、何らかのオペを受ける患者をコホートとして因果推論を行う場合には、交絡因子として解析に投入した方が良いでしょう。
ここで新たに各患者がopeを受けたかどうかを表すope変数、および入院年を表すyearを追加しますので、以下のCodeを走らせてください。
set.seed(777)
hosp_id <- sample(1:10, size = 1000, replace = T)
sex <- sample(0:1, size = 1000, replace = T)
BMI <- round(rnorm(1000, mean = 18, sd = 2), 1)
length_of_stay <- round(rnorm(1000, mean = 30,sd = 10))
ADL <- sample(0:100, size = 1000,replace = T)
smoking_status <- sample(0:100, size = 1000,replace = T)
age <- round(rnorm(1000, mean = 60,sd = 10))
dementia <- sample(0:1, size = 1000,replace = T)
myocardial_infarction <- sample(0:1, size = 1000,replace = T)
liver_disease <- sample(0:1, size = 1000,replace = T)
solid_tumor <- sample(0:1, size = 1000,replace = T)
hospital_bed_size <- sample(20:500,size = 1000, replace = T)
academic_hospital <- sample(0:1, size = 1000, replace = T)
drug_A <- sample(0:1, size = 1000, replace = T)
drug_B <- sample(0:1, size = 1000, replace = T)
death <- sample(0:1, size = 1000, replace = T)
ope <- sample(0:1, size = 1000, replace = T)
year <- sample(2010:2020, size = 1000, replace = T)
df <- data.frame(hosp_id, sex, age, BMI, ADL, smoking_status, dementia, myocardial_infarction,
liver_disease, solid_tumor, drug_A, drug_B, hospital_bed_size, academic_hospital,
length_of_stay, death, ope, year)
df$sex <- as.factor(df$sex)
df$dementia <- as.factor(df$dementia)
df$myocardial_infarction <- as.factor(df$myocardial_infarction)
df$liver_disease <- as.factor(df$liver_disease)
df$solid_tumor <- as.factor(df$solid_tumor)
df$drug_A <- as.factor(df$drug_A)
df$drug_B <- as.factor(df$drug_B)
df$academic_hospital <- as.factor(df$academic_hospital)
df$death <- as.factor(df$death)
df <- prodNA(df, noNA = 0.2)
さて、では病院ごとにオペ回数を合算したhospital volumeを作成しましょう。
Codeにすると、こうです。
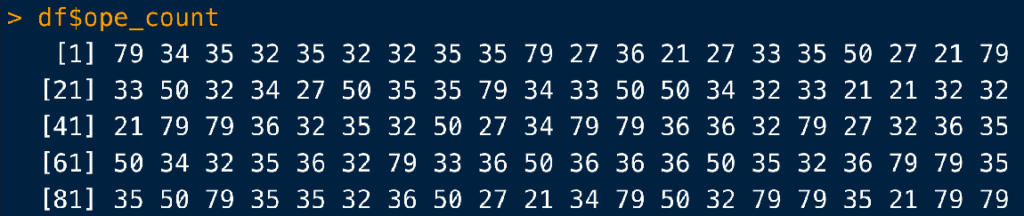
df <- df %>%
group_by(hosp_id) %>%
mutate(ope_count = sum(ope, na.rm = T)) %>%
ungroup()
group_by()で病院を示すhosp_idごとに解析をするように指示を出し、
mutate()でope_countという変数を作成、
さらにその方法として、opeという変数の合計値をカウントするようにsum()で指示を出しています(オペは0/1の整数型の変数ですので、足し算ができます)。
なおその際、いずれかの患者のopeがNAだと、sum()が動きませんので、引数na.rm = TでNAがある患者を無視して合計値を出すように指定しました。
するとこのように、hosp_idごとのope数を合算したhospital volumeが各患者に割り振られます。
なお、hospital volumeは年ごとで変化するため、病院別だけではなく年別に算出してやる方がよく、その場合は以下のように書きます。
df <- df %>%
group_by(hosp_id, year) %>%
mutate(ope_count = sum(ope, na.rm = T)) %>%
ungroup()
このように、group_by()の中に複数の変数を入れてやれば、複数のサブグループで解析を行うことができます。
なお、今回はhospital volumeを作成するのが目的だったため、sum()を使いましたが、group_by()では他にも
mean()
n()
mix()
など様々な関数を組み合わせることができます。
その他の使うかもしれない関数
他にも知っておくと便利な関数があるので、紹介します。
cut_number()は、連続変数を各群の対象者数が等しくなるように分割します。
cut_number(変数, n = 分割数, labels = c(“グループ名1”, “グループ名2”, “グループ名3”))という感じで使います。
cun_interval()は、連続変数を各群の幅が等しくなるように分割します。
rename()は変数名を変更します。
select()は特定の変数のみを抽出します。
たとえば、雑多に作成した変数たちから、最終解析に必要な変数のみを残したい時などに使います。
また、select(-変数名)とすれば、その変数を除外してくれます。
remove()はEnvironmentからデータフレームを削除します。
これくらいかな。
他にも便利な関数は色々あるので、いじりながら覚えていくと良いかなと思います。
終わりに
今日はちょっとボリューミーになっちゃいましたね。
でも、これで解析に必要な変数の作り方を知ることができました!
医療大規模データ解析の中でも、特に時間がかかるフェーズです。
さて、この次は作成した変数に基づき、対象者の包含・除外を行う方法を学びます。
そして、それが終わったらいよいよ解析!!
初心者のRで医療ビッグデータ解析も、ようやく折り返し地点となりました。
ここからは解析結果が見れて、「おお!」と感動するフェーズが多くなりますので、もっともっと楽しく学んで頂けるようになると思います。
一緒にがんばっていきましょー!
すきとほる疫学徒からのお願い
本ブログは、全ての記事をフリーで公開しており、「対価を払ってやってもいいよ」と思ってくださった方のみに、その方が相応しいと思っただけの対価をお支払い頂けるPay What You Want方式を採用しています。
教育に投資できる方だけがさらに知識を身につけ、そうでない方との格差が開いていくという状況は、容認されるべきではないと考えているからです(そもそも私程度の記事によって知識の格差が広がると考えていることが、勘違いかもしれませんが)。
私自身も高校卒業後は大学・大学院の学費、生活費と自分で工面する中で、親の支援を得られる友人たちがバイトをせずに学習に集中したり、海外留学や旅行などの経験を積んだりする様子を見て、非常に悔しい想いをしたことを思い出します。
読者の皆様におかれましては、「勉強になった!」、「次も読みたい!」と本ブログに価値を感じてくださった場合のみ、ご本人の状況が許す限りにおいて、以下のボタンからご自身が感じた価値に見合うだけの寄付を頂戴できますと幸いです。
もちろん価値を感じなかった方、また学生さんなど金銭的に厳しい状況にある方からのご寄付は一切不要です。






