こんにちは、すきとほる疫学徒です。
R Tipsでは、私自身の備忘録も兼ねて、おすすめのRパッケージを紹介します。
80%くらいは備忘録が目的なので、殴り書きになりますことをお許しください。
naniar
naniarは数値による要約、グラフによるVisualizeなど様々な角度からデータの欠損を把握することを助けてくれるパッケージです。
(そんなことはないですが)「なにゃーって読むのかな、かわいいな」とか思ってます。
欠損を把握するパッケージは色々ありますが、naniarはほんとに多種多様なグラフを描いてくれるので、一番のお気に入りです。
数値による要約
まずは、数値による要約から。
miss_var_summary()は、以下のように各変数ごとに生じている欠損の数、割合を算出してくれます。
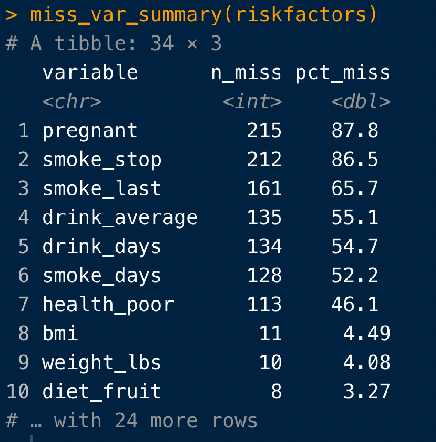
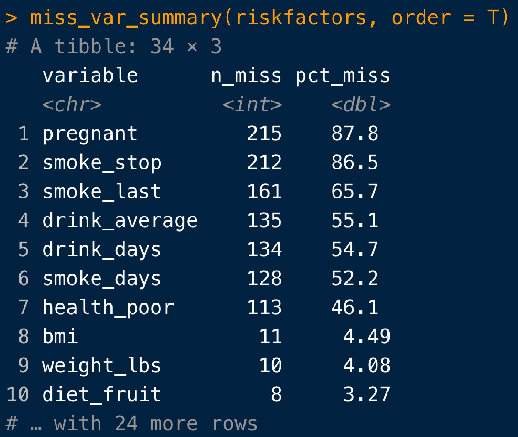
引き数としてorder = Trueをつければ、欠損割合が多い順にソートしてくれます。
欠損の視覚化
ここまでは他のパッケージでも対応できる機能でしょう。
これから説明する欠損の視覚化において、naniarパッケージは特に力を発揮します。
gg_miss_var()
gg_miss_var()は、変数ごとの欠損数をグラフ化します。
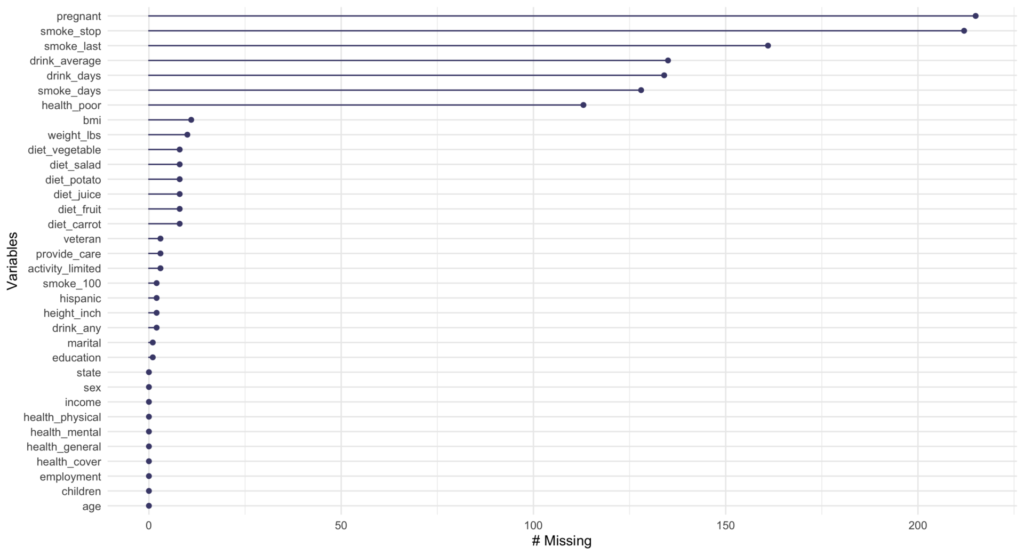
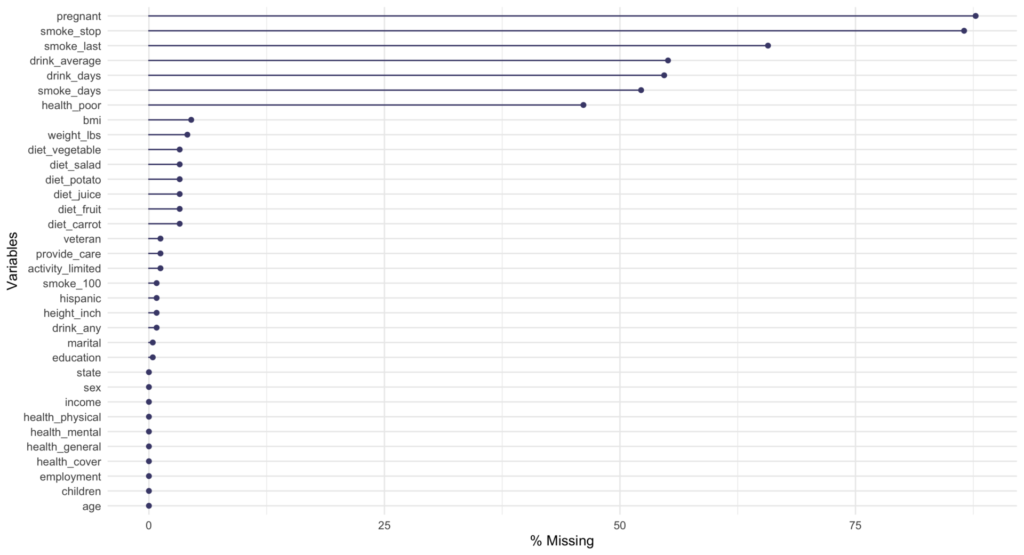
引き数のshow_pct = Tをつければ、欠損数ではなく欠損割合を表示します。
こちらの方が使いやすいですね。
vis_miss_var()
vis_miss_var()も変数ごとの欠損数をグラフ化しますが、同一患者あたりに1ラインが割り当てられるため、変数を超えて欠損がどのように分布しているかを視覚化できます。

ちなみに引き数にcluster = Tをとってやれば、このように欠損をまとめてくれます。
こっちの方が見やすいかな。

geom_miss_point()
お次はgeom_miss_point()。
こちらはggplot()と組み合わせて、点グラフを描画するために使います。
geom_point()で作った通常の点グラフはこちら。
ggplot(airquality,
aes(x = Ozone, y = Solar.R)) +
geom_point()
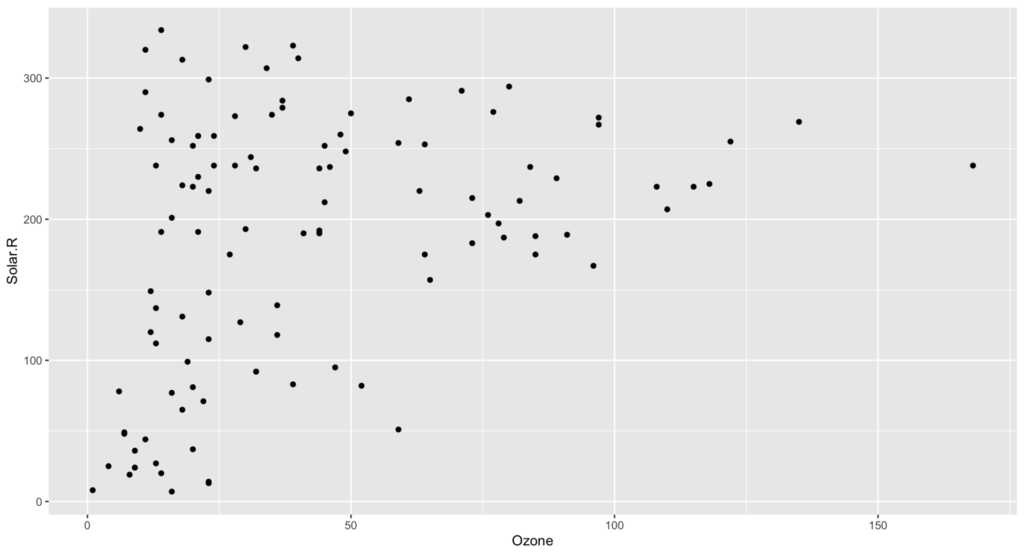
geom_point()では、X軸もしくはY軸に欠損がある場合は除外した上でプロットしています。
それに対し、geom_miss_point()を使うと
ggplot(airquality,
aes(x = Ozone, y = Solar.R)) +
geom_miss_point()
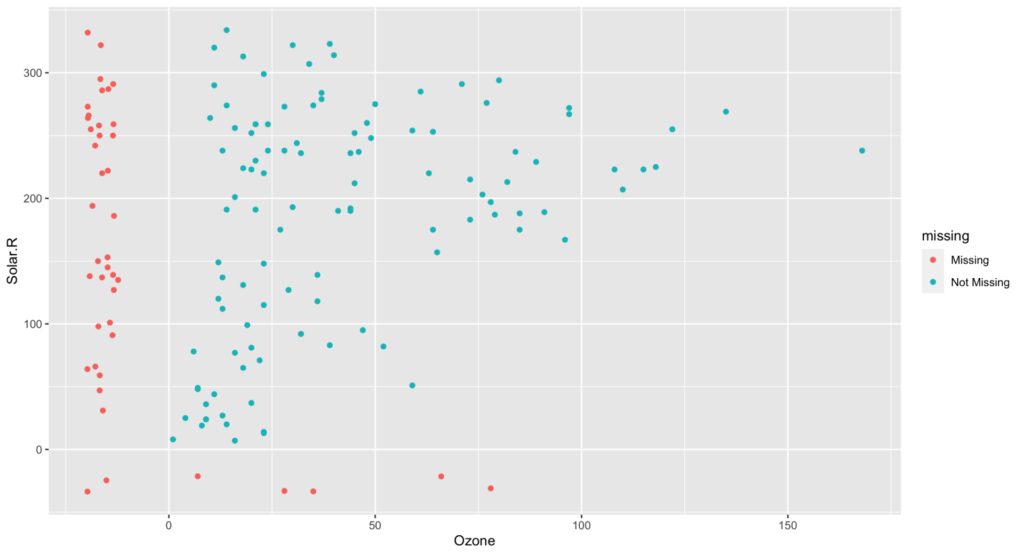
こんな具合に、赤い点でMissingの位置を表示してくれます。
X軸の変数でのmissingはX軸に、Y軸の変数でのmissingはY軸にプロットしていますね。
これを見ることで、各変数の欠損が特定の値近辺に固まっているのか、それとも全体的にバラけているのかなど判断することができます。
gg_miss_upset()
次はgg_miss_upset()。
こちらは、missingの変数間の組み合わせごとにmissingをカウントしてくれます。
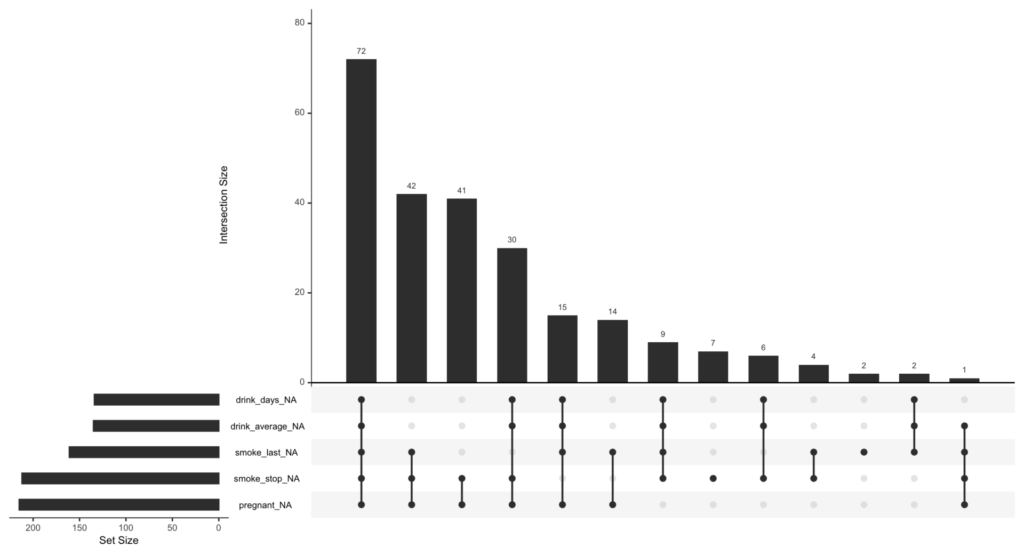
これを見ると、5つの変数全てが欠損しているケースが最も多いということがわかりますね。
特定の変数の組み合わせてmissingが多発していることが発生していれば、そのmissingの背景にある理由を考察することに役立ちます。
mcar_test()
最後はこちら、mcar_test()です。
個人的には、こちらが一番便利だなと感じました。
これは、Missing completely at random (MCAR), Missing at random (MAR), Missing not at random (MNAR)という欠損の3タイプに対して、使っているデータがMCARと仮定した際の仮説を帰無仮説とし、仮説検定をしてくれます。
つまり、MCARが成立しているかどうか(MCARであれば、欠損を除外しても選択バイアスが生じない)をデータで確認することができるということです。
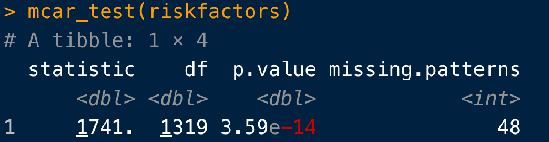
こちらの図のP-valueに該当する数値を見ると、<0.001となっています。
ですので、両側有意水準5%ととすれば、帰無仮説が棄却される、つまりこのデータにおけるmissingはMCARとは言えないということになります。
mcar_test()に関して、欠損データ処理について素晴らしい書籍を出されている長崎大学の高橋先生よりご助言を頂きましたので、引用させて頂きます。
高橋先生のご書籍はこちら
以上で、naniarパッケージの説明は終わりです。
なお、詳細はこちらに記載されていますので、気になる方はどうぞ。
https://cran.r-project.org/web/packages/naniar/naniar.pdf
すきとほる疫学徒からのお願い
本ブログは、読者の方が自由に記事の金額を決められるPay What You Want方式を採用しています。
「勉強になった!」、「次も読みたい!」と本ブログに価値を感じてくださった場合は、以下のボタンをクリックし、ご自身が感じた価値に見合うだけの寄付を頂戴できますと幸いです。
もちろん価値を感じなかった方、また学生さんなど金銭的に厳しい状況にある方からのご寄付は不要です。
引き続き情報発信していく活力になりますので、ぜひお気持ちに反しない範囲でご寄付をお願い致します!








