こんにちは、すきとほる疫学徒です。
R Tipsでは、私自身の備忘録も兼ねて、おすすめのRパッケージを紹介します。
80%くらいは備忘録が目的なので、殴り書きになりますことをお許しください。
1. パイプラインのショートカット
RをRたらしめる機能、そうそれはパイプライン。
おそらく、全世界のRユーザーによる入力頻度No1のコードでしょう。
%>%
そんなパイプラインですが、ショートカットによって一発で入力できることはご存じでしたか?
Mac: Command + Shift + m
Windows: Ctrl + Shift + m
です。
2. 一括ソート
コードを書いていると、書いたり消したりしてるうちに先頭行の位置がばらばらになり、視認性が悪くなってしまうことがあると思います。
そんな時、みなさんどうしてますか?
「一つひとつスペースと削除で直してるよ」
「放置してるよ」
いろんな声が聞こえてきますね。
医療大規模データを解析する際には、残念ながら上で述べたどちらの方法も推奨されないでしょう。
手作業で治すことにより、必要なコードまで気づかずに削除してしまうというヒューマンエラーが起こります。
また、必然的にスクリプトに何行にもわたって多くのコードを書く必要がある医療大規模データ解析では、ざっと見て「何が書いてあるか」を瞬時に確認できるだけの視認性が命です。
なので、放置したままではいけません。
そんな時に役立つのがこのショートカット。
ソートしたい範囲を選択して、
Mac: Command + i
Windows: Ctrl + i
これにより、選択範囲にあるコードを一括で、いい感じにソートしてくれます。
3. 縦に一括選択
Rでは、あるコマンドを使うことで、このように縦に一括選択を行うことができます。
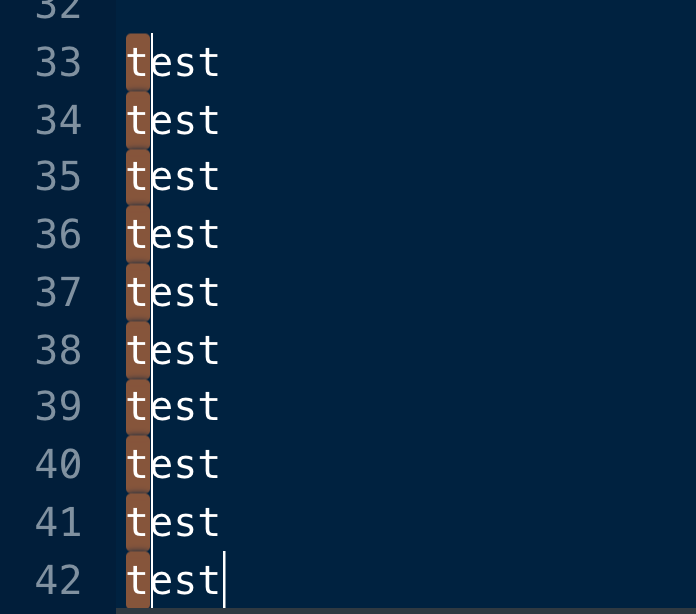
Mac: Option + 選択
Windows: Alt + 選択
です。
そこまで頻繁に使用するわけではありませんが、でも覚えておくと意外と役だったりします。
たとえば、「あー、ここら辺のサブグループ解析は使わないことにしたから、Scriptを走らせた時に反応しないようにしたいな」って時。
#で該当するコード一体をコメントアウトしてあげる必要がありますが、全ての行にいちいち#を入力していくのはとっても面倒です。
そんなとき、該当するコード全てを縦に一括選択し、冒頭にカーソルを合わせて、一度だけ#を入力してやれば、いっぺんにコメントあるとすることができます。
余談ですが、コメントアウトの際は、#をそのまま入力するのでなく、ショートカット
Mac: Command + Shift + c
Windows: Ctrl + Shift + c
を使ってやると便利です。
これをすると、カーソルが行のどこにあったとしても、その先頭に#を入力してコメントアウトしてくれます。
このように、testの後ろにカーソルがあっても、

先頭に#を勝手に入れてくれます。

4. Packageのヘルプを表示
Rでは多種多様なパッケージを使い分け、コードを書いていくことになります。
私もそれなりに頻繁にRを使っていますが、たまに「あれ、このパッケージのこの関数、どんな引数だっけ?」とか、「あれ、あの機能はどの関数だったっけ?」なんてのは日常茶飯事です。
でも、その度にGoogleに飛んで、パッケージ名を入力して、説明記事を開いてなんてしてたら、面倒でしょうがないですよね。
そんな時は、
?パッケージ名
を入力してやれば、Rの画面内にパッケージの解説を開いてくれます。
例えばこちらは、多重代入法を使う際に用いるパッケージ、mice()のヘルプ。

画面上には写っていませんが、この下にもずらーっと丁寧な解説が並んでいて、わざわざGoogle先生に聞かずとも、R上でパッケージ調べを完了することができます。
5. ヘルプのCodeをお試し実行
先ほど紹介したヘルプですが、「いやー、でもCode見るだけじゃ、走らせた時にどんな動きをするかイマイチわかんないんだよね」という声もあるでしょう。
でも大丈夫!!!
なんとそのヘルプの一部、実際に参考例としてCodeを走らせることができちゃいます。
例えば、こちらはggvis()というインタラクティブなグラフを描くためのパッケージのヘルプです(ここでは深入りしませんが、ggvis()ちゃんもとんでもないポテンシャルを秘めたパッケージなので、また改めて詳細を説明します)。
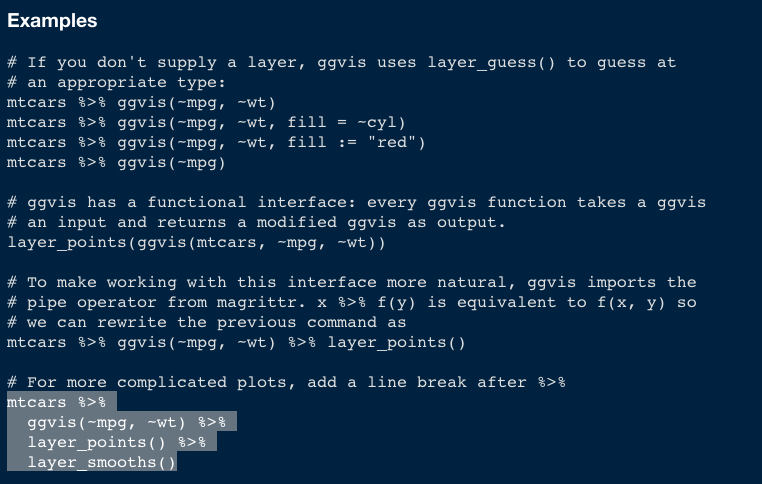
ヘルプの下には、だいたいExampleというCodeの実例が添付されています。
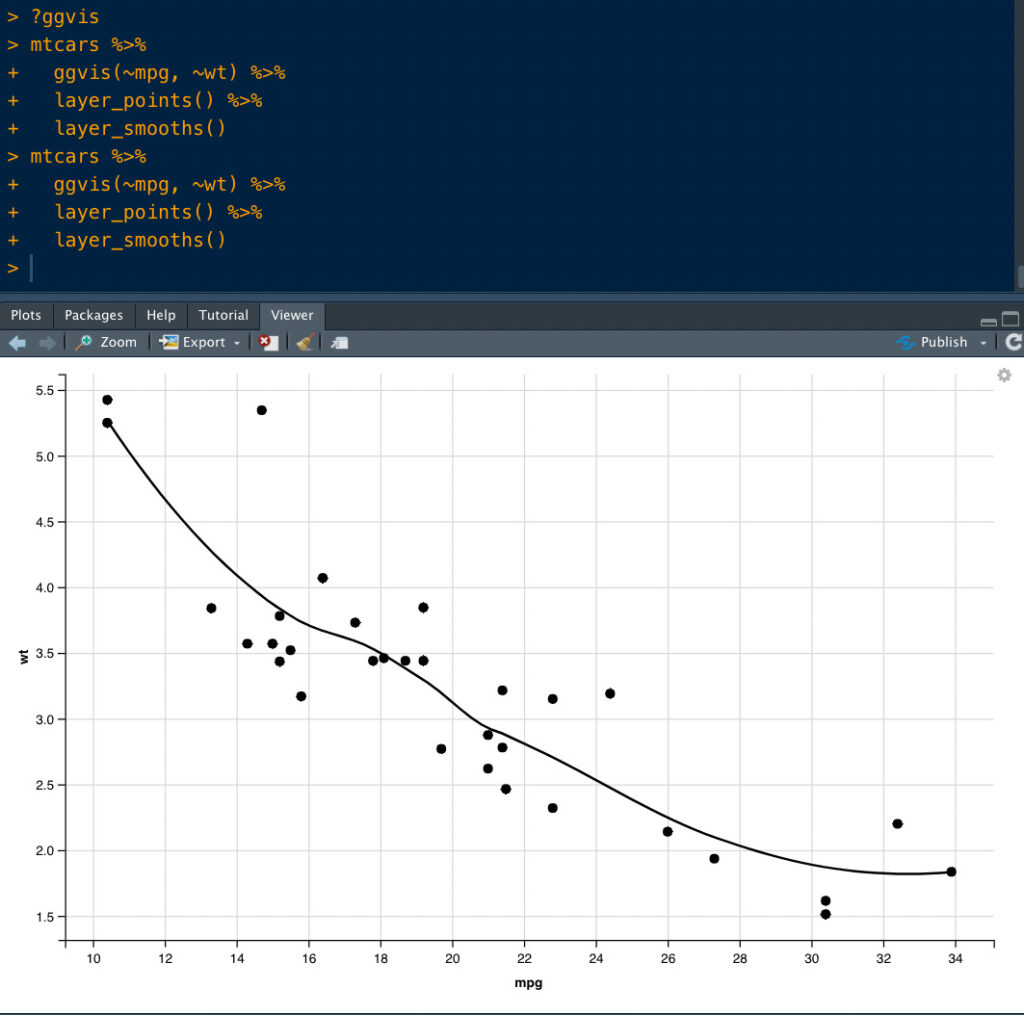
こちらを全選択して、CodeをRunしてあげると、
じゃじゃーーーん!
なんと、実際にCodeを走らせたかのようにシュミレーションしてくれるんですね。
R、あんた、どこまでっ…
未だかつて、こんなに利用者のことを考えてくれる解析ツールが存在しただろうか…
「いやー、それでもまだわかんないよ、もっといい感じの説明ないの?」
さすがにそこまで至れり尽くせりというわけにはいかないので、これ以上のヘルプを求めるのは酷というものです。
諦めて、一つひとつGoogleで探していきましょう。
とでも言うと思ったか!!!
6. browseVignettes()で更なる解説を
Rには、Vignettesと呼ばれるパッケージごとに記載される詳細な取り扱い説明書があります。
Vignettesでは、ヘルプには書ききれないさらに詳細な使用例だったり、グラフだったりを記載してくれています。
browseVignettes(“パッケージ名”)で、そのパッケージのVignettesの一覧に飛ぶことができます。
例えば、
browseVignettes("naniar")
と打てば、自動でブラウザから検索されて、

こんな感じで、Vignettesの一覧が表示されます。
とりあえずHTMLのとこを開いてみればいいでしょう。
ただ、パッケージによってはVignettesが準備されていないものもありますので、その場合はGoogle先生に聞いて自力で調べるしかないでしょう。
7. 折りたたみセクションで視認性向上
RのCodeは、Wordと同じようにセクションを振ることができます。
ショートカットは以下です。
Mac: Command + Shift + r
Windows: Ctrl + Shift + r
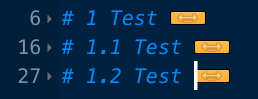
こんな感じで*。
*番号は自動ではつかないので、手動で入力してます。

さらに便利なことに、このセクション、折り畳めちゃうんですよね。
いやー、便利ですね。
医療大規模データを解析していると、長々と同じようなコードを書いていくことがよくあります。
感度分析やサブグループ解析を複数回やると、ちょっとモデルに投入する変数が違うだけで、あとはほぼ同じようなCodeがずらっと並ぶなんてことがよくあるので、一瞬見ただけでは「あれ、あの解析どこだ?」と迷子になっちゃうんですよね。
前もお伝えしましたが、RのCodeはとにかく書き間違えがないように、シンプルかつ分かりやすくする必要があります。
モデルを10個も作ったような際には、論文投稿して6ヶ月後のレビュワー対応のために再度スクリプトを開くと、もう何がなんだか頭のなか大混乱なんてことはよくありました。
Codeを書いているときには、「多少分かりにくくても、このCodeでやってることは絶対忘れない、見れば思い出せる」とか思うかもしれませんが、安心してください、絶対に思い出せません。
「自分を信じない」、これはCodeを書く際の鉄則です。
Codeのダブルチェックをする際にも、セクション別に分かれていて視認性が高かった方が、レビューしてくれる人の負担も大幅に減りますしね。
ですので、このセクション機能は絶対に使った方が良いと思います。
ちなみにこのセクション、Source画面の下にある”#”マークのタブを押せば、自由自在に行き来することができます。
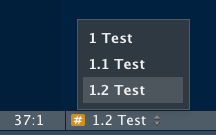
Codeが長くなってくると、「あれ、あのCodeどこいった」とついついスクリプトを上から下まで大冒険してしまいがちになるので、基本はこちらのセクション一覧を使って行き来した方が良いでしょう。
私はあまり使いませんが、
Mac: Command + Shift + o
Windows: Ctrl + Shift + o
でSource画面の右側にもセクション一覧を表示することができます。
8. ファイルを種類別に管理
医療大規模データを解析していると、プロジェクトのフォルダにどんどん色んなファイルが保存されていきます。
オリジナルのCode、オリジナルのデータセットに対して、データセット2以降ができたり、抽出したグラフやエクセルが溜まっていったり。
気づけばフォルダがとんでもない長さになっていたなんてこともよくあります。
となると、
「解析Aの結果なのに、間違って解析Bのエクセルを論文に添付してしまった」
「複数のサブグループ解析の結果が、どのグラフに対応するかわからなくなってしまった」
なんてこともあったりします。
不要なデータセットやグラフを削除しているうちに、削除してはいけないオリジナルのスクリプトやデータセットまで削除してしまったなんてことになれば、最悪ですよね?
そのような事態を避けるために、私は同じプロジェクトでもタイプ別にファイルを管理しています。
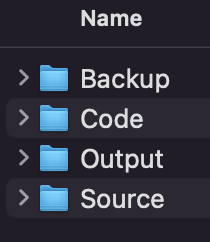
データの読み込みや抽出ごとにパスを変えないといけないのでやや面倒ですが、このおかげでかなりファイル管理がしやすくなりました。
特に、Backupファイルを作るのは強くお勧めです。
私は絶対に削除してはいけないオリジナルのスクリプトとデータセットは、このBackupフォルダにも保存するようにしています。
データセットはそのまま入れたままで、スクリプトの方はその日の作業が終わったときに、Codeファイルに更新された最新のものをコピペするようにしています。
9. TabでCodeを簡単に呼び出し
Rユーザーにとって、最も仲の良いキーボードのボタンはどれか?
それはtabボタンである。
Rの猛者勢であるかどうかは、tabボタンの表記が押し過ぎで擦り切れているかどうかで判別できるとかできないとか…
という冗談はさておき。
パッケージ名や変数名、データセット名を入力するときに、いちいち最初から最後まで入力していたら、面倒なことこの上ないですよね。
そんな時は、冒頭の何文字かを入力した段階で、スッターーーーン!とtabボタンを押してやりましょう。
予測変換してくれます。
さらにこのtabボタン、ありがたいことに関数内の引数も予測で呼び出してくれるんですよね。
これは、mice()という多重代入法を行う関数を呼び出すために、miまで入力してtabボタンを押した上代です。
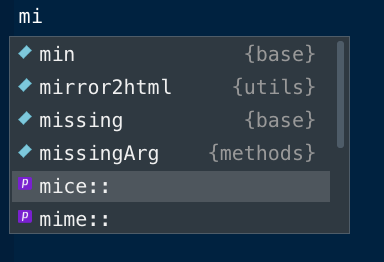
これでパッケージ名のmice::を選択し、さらにその中のmice()関数を見つけるために、miを入力。
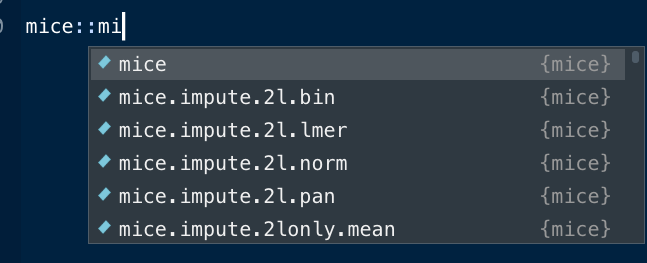
すると、こんな感じでRが「ほら、これが欲しいんだろ?」と言わんばかりにmice()関数を提案してくれますので、選択。
さらにそこから、もう一度tabボタンを押すと、mice()関数の引数一覧がこんな感じで提案されます。
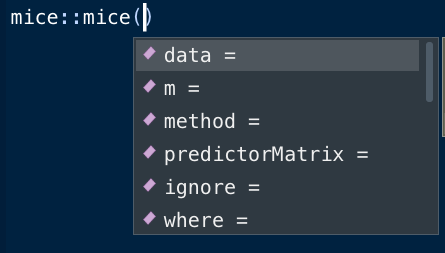
これがマジで楽なんですよ。
引数ってもちろんパッケージごとに違うので、「あれ、この関数の引数はなんだったかな?」と記憶喪失になることも日常上茶飯事なのですが、こんな感じで引数を出してもらえると、「あー、はいはい」と思い出せます。
何より、入力が楽。
こいつめっちゃ賢くて、上の引数から一つを選択して、入力し、再びtabボタンを押すと、残りの引数一覧をまた出してくれるんですよね。
しかも、一度入力された引数は除外して表示してくれるから、ダブって入れる心配はなし。
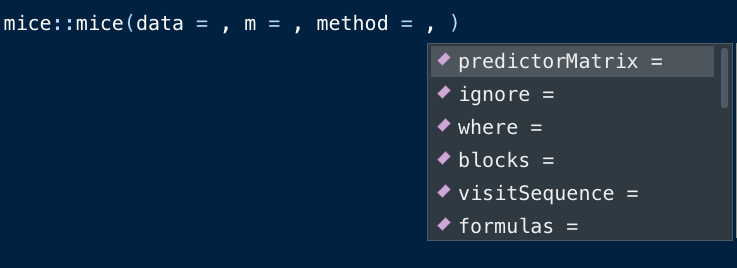
「tabボタン使ってなかったよー」って人は、騙されたと思って使ってみてください。
あまりの楽さにR生活が激変するはずです。
10. 日付データを一瞬で入れる
医療大規模データの解析は、短くて数週間、長いときには数ヶ月かかることもあると思います。
日を跨いで作業していると、「作業日を入れておこう」みたいな気持ちになることもあるかもしれません。
そんな時も、とあるショートカットを使えば、一瞬でコメントアウトした作業日を差し込むことができます。
Mac: ts + tab + Enter
Windows: ts + tab + Enter
これだけで、いちいち年月日時を入れる手間が省けます。
11. 回帰分析の結果を綺麗にTableにする
みなさん、Rで行った回帰分析の結果、いつもどうやって論文投稿用のTableに変換していますか?
私はいつも、write.csvで抽出して、その後はエクセルで体裁を整えています。
ただ、論文投稿時はともかく、共著者の方にチェック頂くたびに、こうしてTableを整えているとかなり時間が消費されちゃうんですよね。
せっかく1時間くらいかかって関連するTableを整えても、共著者チェックで「この変数も追加したら?」、「対象者の除外基準はこうしたら?」と助言を頂いて、解析やり直しなんて当然のようにあるので、Tableを整えることごときでそんなに時間を使いたくない、というのが正直なところだと思います。
だからといって、write.csvで吐き出された乱雑なエクセルをそのままお見せするのも失礼ですし。
そんな時、私はこちらのパッケージでサクッと見た目の良いTableを作ることにしています。
model <- lm(data = iris,
Sepal.Length ~ Sepal.Width + Petal.Length + Petal.Width + Species)
install.packages("parameters")
install.packages("gt")
library(parameters)
library(gt)
print_html(model_parameters(model,
summary = T))
こうすると、自動でこんな感じに見目麗しいTableを吐き出してくれます。
引数のsummary = Tにすると、ご覧の通りTableの下にモデルを添付してくれます。
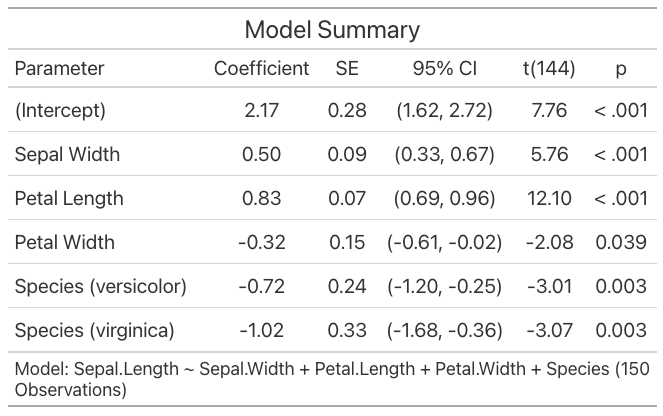
これを知ってから、「あー、どうせ解析やり直しになるのに、なんでこんな手間かけてTable作ってんだ」という不満から解消されたので、”parameters”パッケージには本当に感謝しています。
12. 行ごと削除するショートカット
Mac: Command + d
Windows: Ctrl + d
で行ごと一括削除できます。
終わりに
いかがでしたでしょうか?
Rは無限の可能性を秘めているので、きっとこの世には私の知らないTipsも存在するはず。
ブログに追記しますので、みなさんも「これは凄いぞ!」というとっておきTipsがありましたら、ぜひぜひ教えてくださいませ。
なお、こちらの記事では研究者が勧める研究お役立ち本を紹介しています。
論文の書き方に始まり、思考法、メンタルケア、そして研究者キャリアなど、さまざまな角度からみなさんの研究者生活を支援する『神本』たちを紹介していますので、ぜひご覧ください。
すきとほる疫学徒からのお願い
本ブログは、読者の方が自由に記事の金額を決められるPay What You Want方式を採用しています。
「勉強になった!」、「次も読みたい!」と本ブログに価値を感じてくださった場合は、以下のボタンをクリックし、ご自身が感じた価値に見合うだけの寄付を頂戴できますと幸いです。
もちろん価値を感じなかった方、また学生さんなど金銭的に厳しい状況にある方からのご寄付は不要です。
引き続き情報発信していく活力になりますので、ぜひお気持ちに反しない範囲でご寄付をお願い致します!








