こんにちは、すきとほる疫学徒です。
【初心者のためのRで医療ビックデータ解析】シリーズは、「これまでデータの解析を行ったことがない人」を対象にして、Rで医療ビックデータを解析する方法をわかりやすく解説していきます。
全てのシリーズを受講し終えた時点で、「診療報酬データやレジストリーといった医療大規模データを、自立して解析できる」状態になることを目指しております。
全研究者、Rユーザー化計画、ここに始動す。
こちらの記事はパート4になりますので、まずそれ以前のパートをお読み頂いてから、この先に進んで頂けますと、より一層Rの勉強が楽しめるかと思われます。
Projectを作成しよう
Projectとは、R特有のファイル管理方法でして、Projectに紐づくR Studioの設定や、ファイル、ワーキングディレクトリなどを一括で管理してくれます。
要は、「そのプロジェクトに関連した全ての物事をまとめ管理できる仕組み」のようなものだと思ってください。
ですので、まずR Studioを立ち上げて、このProjectフォルダを作成しましょう。
その前に、Projectフォルダの格納先として、デスクトップにR_fileというタイトルでフォルダを作っておいてください。
フォルダが作成できたら、いよいよR Studioを開きましょう。
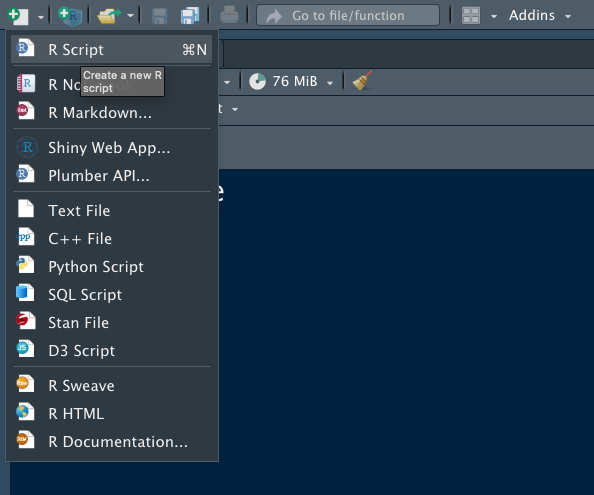
左上のタブの”+R”と書かれたアイコンをクリックします。
すると、以下のようなポップアップが出現します。
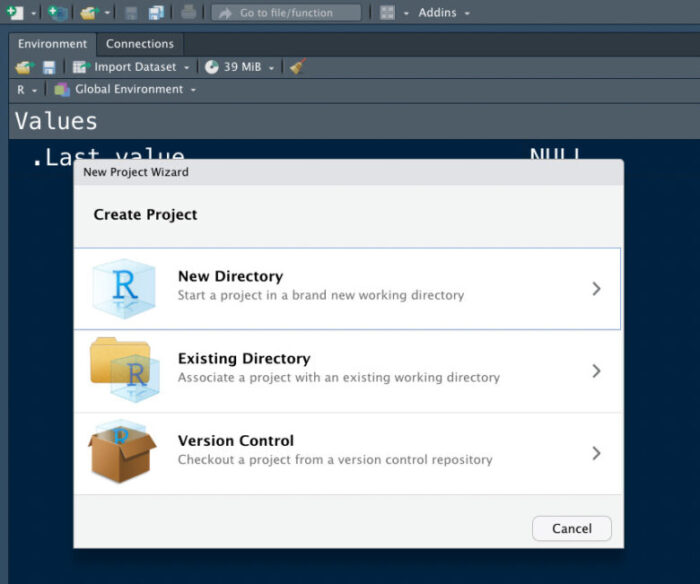
そしたら、New Directoryを選択です。
下のような画面になりますので、New Projectを選択しましょう。
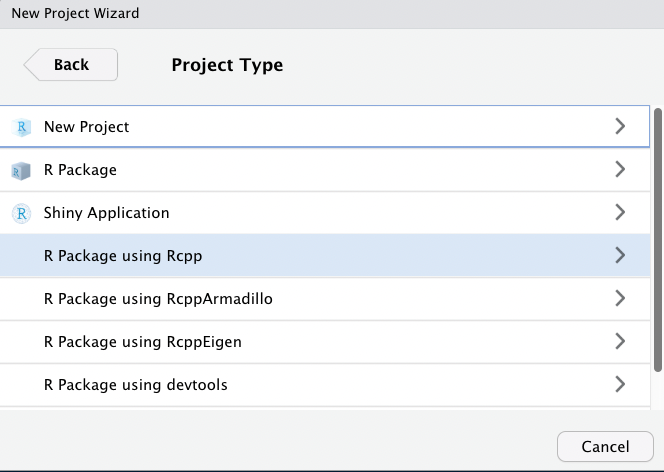
そうすると、このような画面になります。
Directory nameは何でもいいですが、とりあえずR_trainingにしておきましょう。
また、その下のBrowseというボタンを押して、Projectフォルダの保存先として先ほど作成したデスクトップのR_fileを指定しましょう。
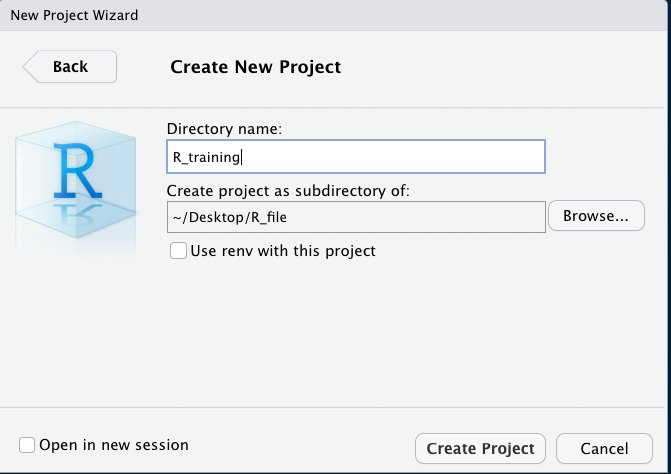
そうすると、自動でR Studioのメイン画面に切り替わり、Projectが作成されているはずです。
ここでは、次回からどのようにファイルを開けば良いのかを学ぶため、一旦R Studioをクローズしてください。
閉じたら、デスクトップのR_fileを開きましょう。
すると、中に先ほど作成したR_trainingというフォルダが作成されているはずです。
そのフォルダを開くと、中にR_training.Rprojという名前でProjectフォルダが作成されていることが確認できたはずです。
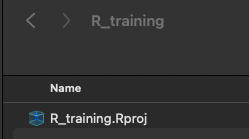
ですので、次回から作業を始めるときには、このProjectフォルダからR Studioを立ち上げるようにしましょう。
ちなみに、Projectフォルダが正しく開けていると、画面右側に以下のようにProject名が記載されます。
では、Projectフォルダを開いていただき。
Codeを書く場所として、Scriptを展開します。
左上のアイコンをクリックしてもいいでが、ショートカットの方が楽なので、ショートカットで開く癖をつけると良いでしょう。
ショートカットはこちらで紹介しております!
以上が、新たにScriptを開く方法でしたが、保存する際にはこのフロッピーマークを押しましょう。
すると、同じProjectフォルダの中にファイルが保存されているはずです。
保存したScriptを再度開いて作業を再開する際には、必ずProjectを立ち上げてから開くようにしましょう。
Projectの中でScriptを立ち上げないと、そのProjectで設定しているパスにScriptが紐づかなくなってしまいます(パスの説明は次のチャプターで)。
パスを設定しよう
これから、Rを使う上での住所の役割を果たすパスについて解説します。
なお、Projectを使用している場合には、そのProjectがあるフォルダに自動的にパスが設定されますので、手動でのパスの設定は基本的に不要です。
ただ、私の場合はProjectフォルダ内でさらにOutput、Source、Code、Backupというようにファイルごとに分類しているので、保存先に合わせて都度ファイルを設定するようにしています。
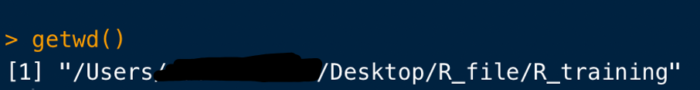
まずパスですが、以下のコードで現在のパス、つまりどこの住所にR Studioが紐づいているかを確認できます。
getwd()
すると、Console画面にこんな感じでパスが表示されます。
新たにパスを設定したい場合には、
setwd()
で、()の中にパスを入力すれば設定できます。
住所として設定したいフォルダのパスを知るには、設定したいフォルダを開いて右クリック、そして
Windows:フォルダマークをクリック
Mac:フォルダを右クリック、optionを押すと”パスをコピー”というコマンドがでます。
今回は、Projectフォルダの住所に既にパスが設定されているので、新たにパスを設定する必要はありません。
ちなみに、パスをコピー&ペーストすると、”/”のところが、”¥”だったり”\”になっていることがあります。
これだとRがパスをうまく認識してくれませんので、手作業で”/”に変えていくようにしましょう。
Windowsだと、”¥”になる気がします。
Packageをインストール/ロードしよう
以前もお伝えしましたが、Rの魅力は様々なスキルがセットされた技マシンであるPackageを使える点にあります。
今後、必要な解析が登場するたびにPackageをインストールして頂きますが、頻繁に使うパッケージは最初にインストールしてしまいましょう。
install.packages("tidyverse")
install.packages("lubridate")
install.packages("haven")
install.packages("summarytools")
install.packages("ggplot2")
install.packages("tableone")
install.packages("dplyr")
install.packages("naniar")
install.packages("devtools")
install.packages("reader")
install.packages("missForest")
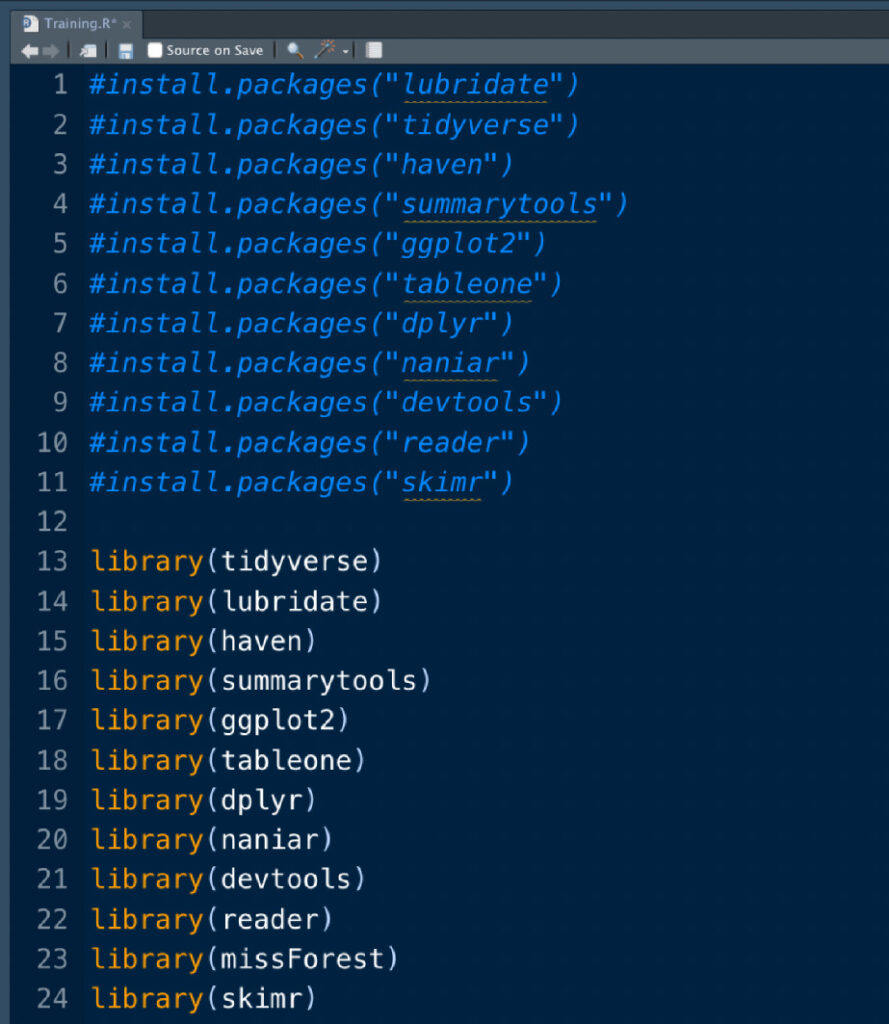
library(tidyverse)
library(lubridate)
library(haven)
library(summarytools)
library(ggplot2)
library(tableone)
library(dplyr)
library(naniar)
library(devtools)
library(reader)
library(missForest)
install.packages()はパッケージをオンラインからダウンロードするための関数です。
library()はダウンロードしたパッケージをアクティベートするための関数です。
install.packages()は一度すればもう行う必要はありませんが、library()に関しては毎回スクリプトを立ち上げるためびに行う必要があります。
どのプロジェクトでも、ほとんどのパッケージは共通して使うことになるため、パソコンのメモ帳みたいなところにlibrary(パッケージ名)の一覧を書いておいて、都度コピペできるようにしておくと、今後の作業が楽になると思います。
また、Scriptを実行するたびに間違ってinstall.packages()までRunしてしまうと、いちいちRから「このパッケージもうインストールされてるけど、本当にやる?」と聞かれて面倒なので、一度パソコンにインストールしたあとは、install.packages()は#でコメントアウト(Runの対象にならないようにするマーク)しておきましょう。
コメントアウトのショートカットはこちらで紹介しています。
こんな感じね。
今回は一括でインストール、アクティベートしましたが、それぞれのパッケージの内容は、時間があるときにぜひ調べておいてください。
これらは、今後何度も使うことになる重要なパッケージです。
ファイルを読み込もう
さて、これで解析を行うための下準備が整いましたので、いよいよファイルを読み込みましょう。
と言いたいところですが、読み込むためには皆さんの手元に今回の解析で使うファイルがないといけません。
ですので、代わりに以下のCodeを入力して頂き、解析の対象となるデータセットを作成して頂きます。
こちらのコードを入力し、Runしてください(Runをショートカット入力する方法は過去記事で紹介しています。)。
# Dataset -----------------------------------------------------------------
set.seed(777)
hosp_id <- sample(1:10, size = 1000, replace = T)
sex <- sample(0:1, size = 1000, replace = T)
BMI <- round(rnorm(1000, mean = 18, sd = 2), 1)
length_of_stay <- round(rnorm(1000, mean = 30,sd = 10))
ADL <- sample(0:100, size = 1000,replace = T)
smoking_status <- sample(0:100, size = 1000,replace = T)
age <- round(rnorm(1000, mean = 60,sd = 10))
dementia <- sample(0:1, size = 1000,replace = T)
myocardial_infarction <- sample(0:1, size = 1000,replace = T)
diabetes_mellitus <- sample(0:1, size = 1000,replace = T)
liver_disease <- sample(0:1, size = 1000,replace = T)
solid_tumor <- sample(0:1, size = 1000,replace = T)
hospital_bed_size <- sample(20:500,size = 1000, replace = T)
academic_hospital <- sample(0:1, size = 1000, replace = T)
drug_A <- sample(0:1, size = 1000, replace = T)
drug_B <- sample(0:1, size = 1000, replace = T)
death <- sample(0:1, size = 1000, replace = T)
df <- data.frame(hosp_id, sex, age, BMI, ADL, smoking_status, dementia, myocardial_infarction, diabetes_mellitus,
diabetes_mellitus, liver_disease, solid_tumor, drug_A, drug_B, hospital_bed_size, academic_hospital,
length_of_stay, death)
df <- prodNA(df, noNA = 0.2)
df$sex <- as.factor(df$sex)
df$dementia <- as.factor(df$dementia)
df$myocardial_infarction <- as.factor(df$myocardial_infarction)
df$diabetes_mellitus <- as.factor(df$diabetes_mellitus)
df$liver_disease <- as.factor(df$liver_disease)
df$solid_tumor <- as.factor(df$solid_tumor)
df$drug_A <- as.factor(df$drug_A)
df$drug_B <- as.factor(df$drug_B)
df$academic_hospital <- as.factor(df$academic_hospital)
df$death <- as.factor(df$death)
これで、dfという名のデータセットが作成されました。
皆さんの画面で、”Environment”というパネルにdfが表示されているはずです。
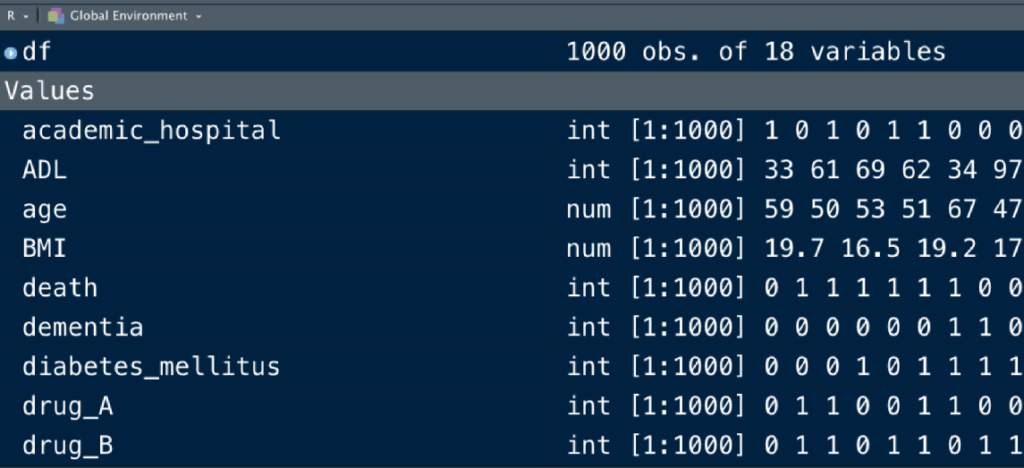
もしdfを修正してdf1、df2を作っていった場合には、dfの下に新たに作成したデータセットが並んでいくことになります。
ここがRの強みの一つです。
SPSSのように、一つのデータセットに対して解析を行っていく解析ツールだと、Rのように複数のデータセットを瞬時に行き来することができないんですよね。
Rなら、コード上で対象とするデータセットを指定してあげるだけで、同じスクリプトの中でいくつでもデータセットを扱うことができるので、非常に解析がスムーズになります。
なお、パソコン上に読み込みたいcsvファイルがある際は、以下のようにすると読み込んでくれます。
df <- read_csv("パス名")
パス名は、読み込みたいcsvファイルのパス名を入手して、入力してください。
その際、パスの繋ぎ目が”/”になっていることを確認しましょう。
<-は、=と同じニュアンスの記号です。
ただファイルを読み込むだけだと、データセットとして保存してくれませんので、”df <-“は、読みこんだファイルをdfという名前のデータセットとして保存してくださいね、という指示を出しています。
ちなみに、同じような関数にread.csv()がありますが、こいつはフェイクです(フェイクじゃないんですけど)。
どちらもcsvファイルを読み込んでくれますが、read_csv()の方が早く、良い感じに読み込んでくれるので、間違いませんようお気をつけください。
他の形式のファイルを読み込む場合は
SPSS: read_spss()
Stata: read_dta()
SAS: read_sas()
などです。
これらは、先にインストールしているhavenというパッケージに含まれた関数です。
havenをインストール、アクティベートしていないと使えませんので、お気をつけください。
ファイルを読み込んだ際に文字化けが発生している場合は、ファイルのエンコーディングを正しく読み取れていない可能性があります。
こちらの記事を参考に、R StudioのエンコーディングをUTF-8に設定した上で、読み込んでみてください。
それでも読み込めない場合は、まずはファイルのエンコーディングを調べましょう。
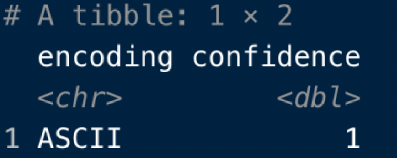
guess_encoding("ファイルのパス名")
です。
すると、こんな感じでファイルのエンコーディングを返してくれます。
そしたら、エンコーディングを指定してファイルを読み込むので、
df <- read_csv("ファイルのパス名", locale = locale(encoding = "エンコーディング名"))
です。
ただ、私自身はこれまで”UTF-8″で正しく読み込めなかったファイルに遭遇したことがないので、医療大規模データ研究をしている限りは、上で書いたコードを使う状況はあまりないような気がします。
データを観察しよう
データを手にしたらまずすべきこと、それはデータの観察です。
あの角度、この角度からとデータをじっくりと眺めて、全体像を把握していきましょう。
最も簡単なデータの観察方法は、Environmentに表示されたdfのボタンを押すことです。
クリックしますと、こんな感じで新たなタブが立ち上がり、それぞれの変数とその値を示してくれます。
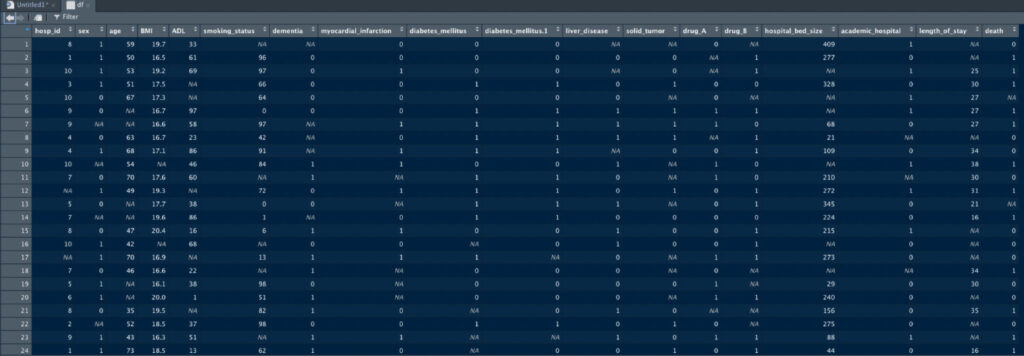
なお、今回作成したこのデータセットは、1行あたりが各患者の一入院ごとのエピソードになっており、そのエピソードにおける年齢、性別、ADL、喫煙歴などの情報が含まれています。
より複雑になってくると、同じ患者が複数の行を持ったり、複数のシートにデータがばらけているなどがあるのですが、このセミナーでは触れません。
このデータテーブルは、観察のために直接操作することができます。
まず、各変数の隣にある矢印を押してあげると、昇順・降順それぞれに行を並び替えることができます。
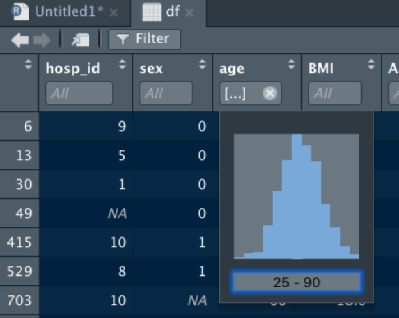
また、Filterボタンを押せば、このように各変数ごとにフィルタリングすることができます。
ちなみに、データセット名をクリックせずとも、以下のコードでこちらのTableは呼び出し可能です。
View(df)
データを要約するパッケージを使おう
さて、上のTableを使って十分にデータが観察できたかというと、そうではないですよね?
ただ変数ごとに数字の羅列を眺めているだけなので、集団の傾向などは全くわかりません。
というわけで、便利なパッケージを使いながらより詳しくデータを観察していきましょう。
glimpse()
まずは
glimpse(df)
その名の通り、データの型と冒頭の数行を「チラッと見る」ための関数です。
View()でTable全体を開くと、データの容量によっては数分かかることもあるため、「はーん、こういう変数があって、こんな感じで入力されてんのね」と本当に触りを掴みたいときに使用すると良いでしょう。
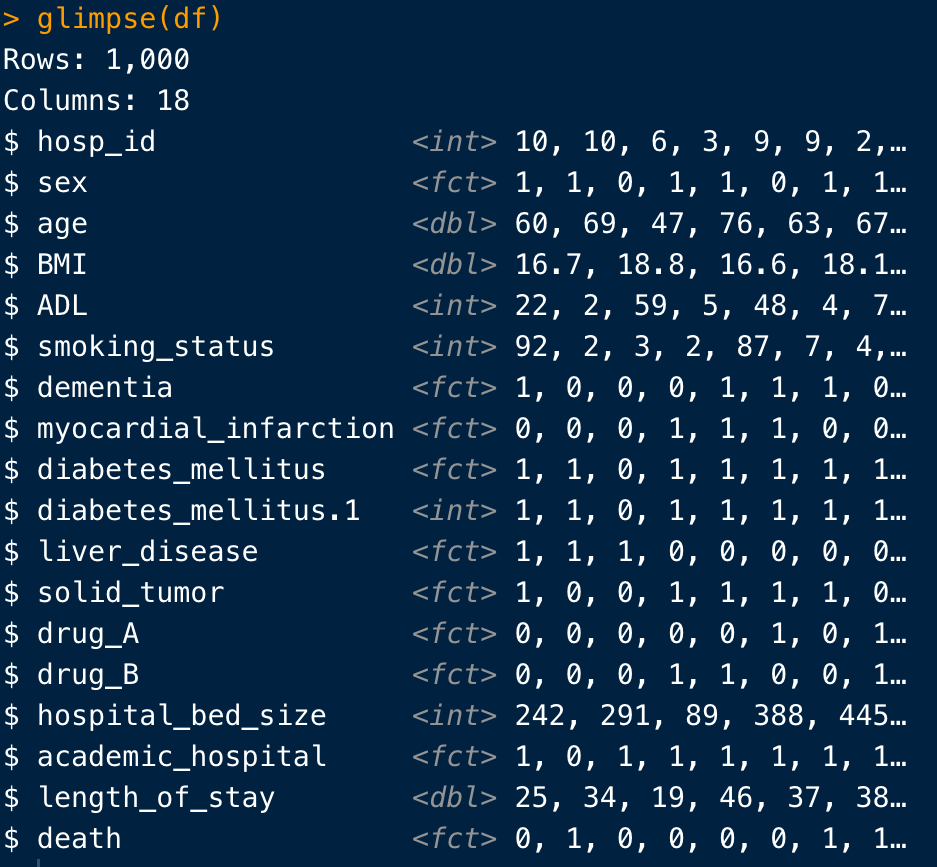
ちなみにRにはデータの型という概念があり、それぞれの変数の属性?のようなものを表しています。
characterは文字列
integerは整数
doubleは実数
numericは整数と実数
factorは順序なし因子(カテゴリー変数はこれ)
などです。
関数によっては特定の型にしか対応していないものもありますし、また型が違うと各関数でのアウトプットも変わってきます。
例えば、年齢を連続値とするか、カテゴリー変数とするかで、表の表記が変わりますよ(連続変数なら最大、最大値などの要約統計量が表示されますが、カテゴリー変数だと各年齢ごとに患者数(割合)が表示され、見にくいことこの上なしです)。
summary()
次は
summary(df)
これは、このように連続変数は最小値、最大値の要約値、カテゴリー変数は割合を算出します。
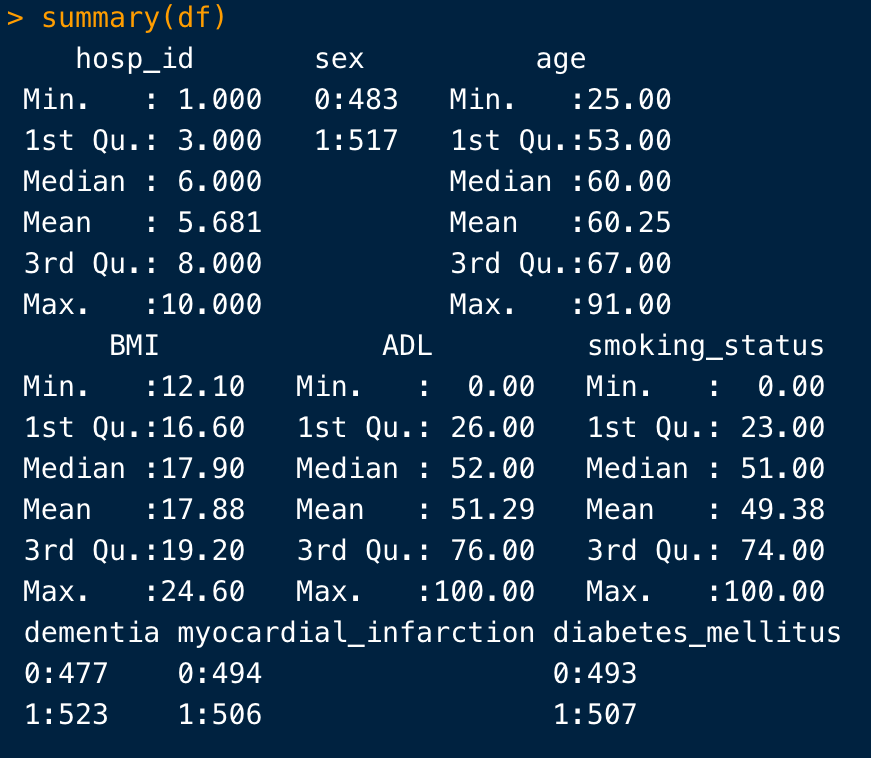
このように、Rの関数は変数の方に応じて処理方法が変わるため、どの型で変数を扱うかはとても大切になります。
「あれ、解析が回らない」という際の原因として、「その関数が対応していない型で変数を投入していた」ということが頻繁に起こるので、注意しましょう。
なお、データセット全体ではなく特定の変数の要約統計量だけを出したい場合には、
summary(df$age)
のように、データセット名の後に$で繋いで、変数名を指定します。
$は多用しますので、覚えておきましょう。
table()
次は
table()
連続変数は無視して、カテゴリー変数の割合を算出したい場合は、table()関数を使用します。
table(df$変数)とすれば、各カテゴリーごとの患者数をカウントすることができます。
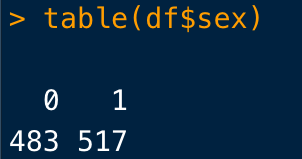
割合にしたいときには、
prop.table()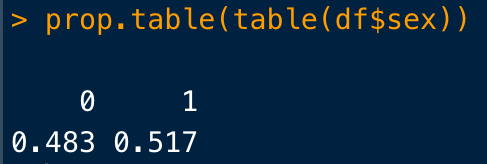
2×2表を作りたいときには、変数を2つ入れればOKです(両方カテゴリー変数でないといけません)
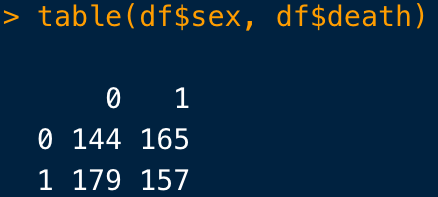
table()は単純に、「男女でアウトカムを発症してる人って、そもそも何人?」みたいなことを瞬間的に知りたいときに役立ちます。
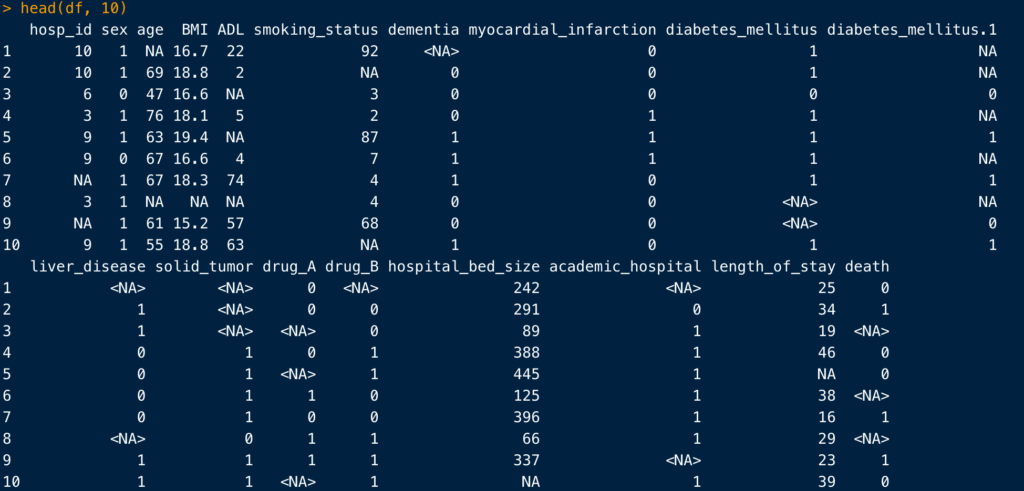
head()
あまり出番はありませんが、head()は各変数の冒頭の数行(だからheadですね)を表示してくれます。
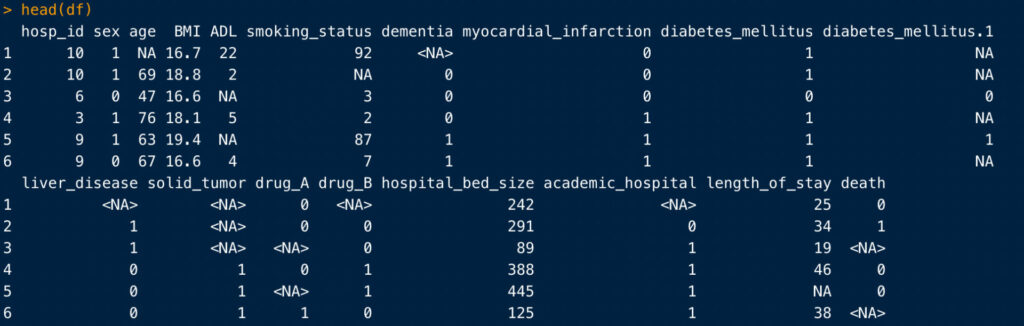
head(df, 10)のようにすると、表示する行数を指定できます。
skim()
ここで新たな関数を紹介しましょう。
まず、skimrというパッケージをインストール、アクティベートしてください。
どうやるんでしたっけ?
正解は、
install.packages("skimr")
library(skimr)ですね。
実行したら、install.packages()は忘れずにコメントアウトしましょう。
一度インストールしたら、2回目以降は不要ですので。
なお医療大規模データを解析していると、進めるにつれて「あ、ここは新しくあのパッケージを使ってみよう」みたいに解析の途中で新キャラが登場することが多々あります。
そういう時も、install.packages()とlibrary()は必ずScriptの冒頭まで戻って、そこに追加する形で書くようにすると良いです。
Scriptを開いた際に、まずやることはその解析で使用するパッケージを一括でアクティベートすることですので、冒頭にパッケージ群が並んでいた方が楽なんですよね。
また、Scriptの冒頭を見れば「あ、あのパッケージ忘れてたわ」なんていうふうにも気づくことができます。
さて、今回使用するのはskimrパッケージより、skim関数です。
skim(df)
実行すると、このように変数のoverviewが表示され、
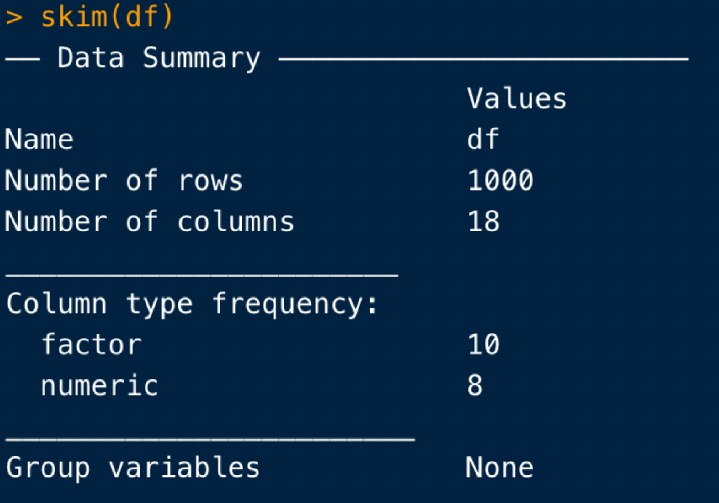
その下に、カテゴリー変数と連続変数別のサマリーが並びます。
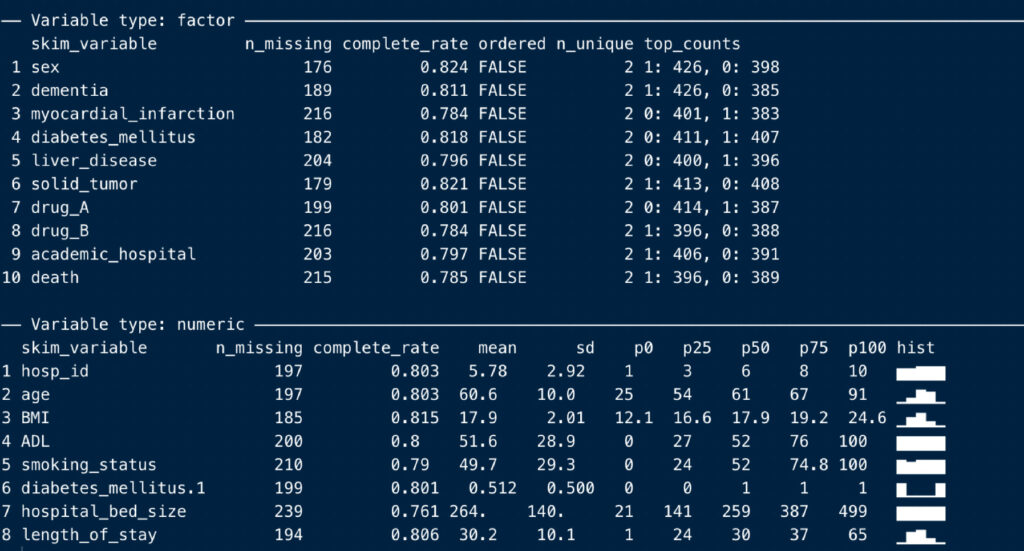
n_missing: 欠損数
complete_rate: 非欠損の割合
n_unique: カテゴリー数
top_counts: 各カテゴリーの対象者数
などですね。
ちょっと気が利いている点として、連続変数には右端にヒストグラムが追加されます。
dfSummary()
skimに似た関数として、dfSummaryがあります。
dfSummary(df)
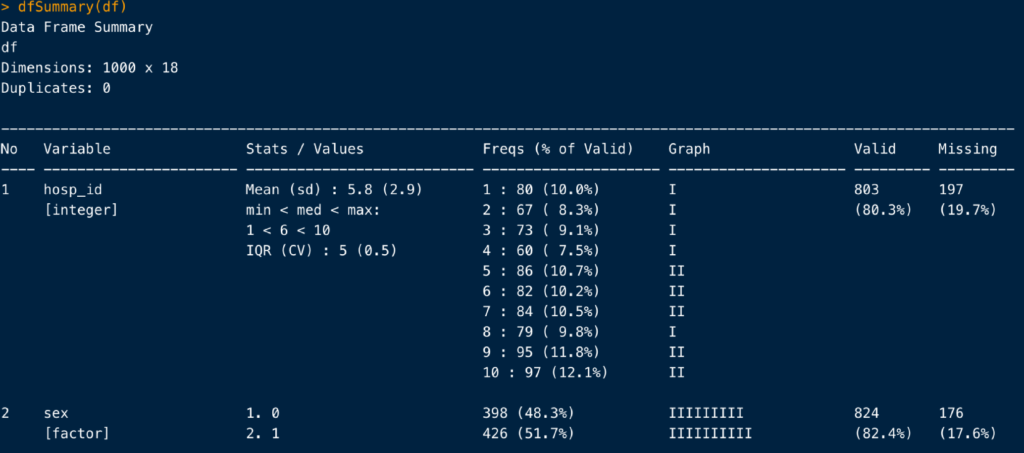
こんな感じで、skim()と似たような結果を返してくれます。
でも、skim()もdfSummary()も、ちょっと見にくいですよね。
そんな時はこれ!
こちらが、私が一番愛用しているデータの記述方法です。
view(dfSummary(df))
そうすると、どーん(画像が荒いのはお許しください)!!!
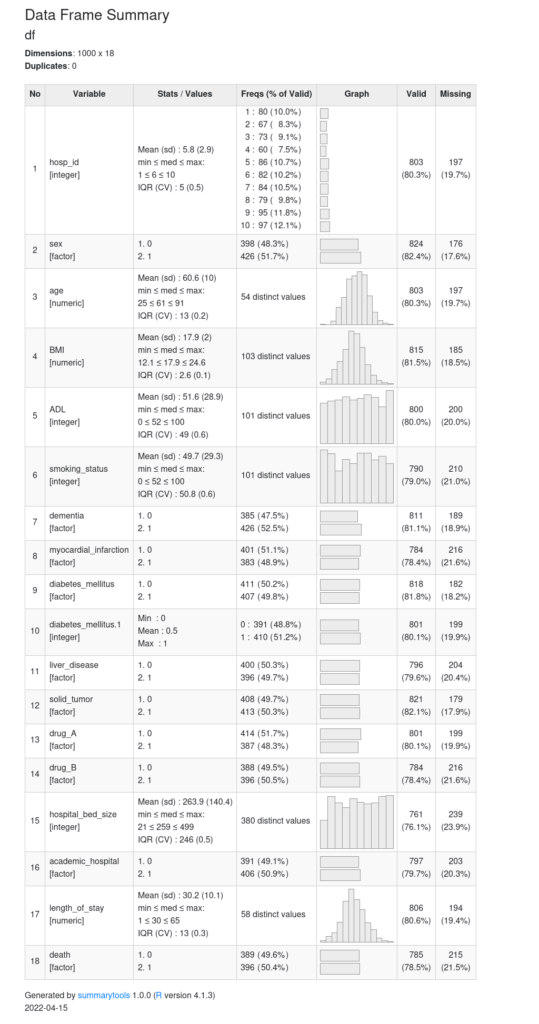
綺麗!
わかりやすい!
というわけで、個人的にはRでのデータ要約には一番こいつがおすすめです。
CreateTableOne()
もう一つ、必要不可欠な関数を紹介します。
CreateTableOne()
こちらでは、量的研究の論文では間違いなく掲載されるTable1(対象者の属性の要約)を、良い感じに出力してくれます。
これはRで医療大規模データ解析をやるからには必須とも言える関数であり、絶対に使った方が良いです。
長くなるため、詳細はこちらの記事で書いておりますので、ご覧ください。
欠損の把握
医療大規模データ解析においては、存在するデータを把握することと同じように、欠損の把握も非常に重要になります。
欠損の把握方法はこちらの記事に書いてありますので、ぜひ参考になさってください。
終わりに
以上になります。
基本的には、今回紹介したパッケージがあれば医療大規模データの観察には十分だと思いますが、Rには同じように記述統計を把握することができるパッケージがまだまだ存在します。
皆さんのお気に入りのパッケージを見つけて、医療大規模データ解析の相棒にしてみるのも良いかもしれませんね。
さて、データの観察が終わりました。
次回はいよいよデータの加工、変数の作成方法を学んでいきましょう。
すきとほる疫学徒からのお願い
本ブログは、全ての記事をフリーで公開しており、「対価を払ってやってもいいよ」と思ってくださった方のみに、その方が相応しいと思っただけの対価をお支払い頂けるPay What You Want方式を採用しています。
教育に投資できる方だけがさらに知識を身につけ、そうでない方との格差が開いていくという状況は、容認されるべきではないと考えているからです(そもそも私程度の記事によって知識の格差が広がると考えていることが、勘違いかもしれませんが)。
私自身も高校卒業後は大学・大学院の学費、生活費と自分で工面する中で、親の支援を得られる友人たちがバイトをせずに学習に集中したり、海外留学や旅行などの経験を積んだりする様子を見て、非常に悔しい想いをしたことを思い出します。
読者の皆様におかれましては、「勉強になった!」、「次も読みたい!」と本ブログに価値を感じてくださった場合のみ、ご本人の状況が許す限りにおいて、以下のボタンからご自身が感じた価値に見合うだけの寄付を頂戴できますと幸いです。
もちろん価値を感じなかった方、また学生さんなど金銭的に厳しい状況にある方からのご寄付は一切不要です。







